Посещений: 
РИБОСОМЫ
Сборка
Ribosome assembly coming into focus • Sebastian Klinge &
• John L. Woolford Jr
 Nature Reviews Molecular Cell Biology volume 20, pages116–131 (2019) |
| |
|
Синтез белков у эукариот осуществляется с помощью рибосом, крупных РНК-белковых комплексов, состоящих из малой и большой субъединиц. Во время белкового синтеза декодировение мРНК осуществляется с помощью малой субъединицы, это сопровождается формированием пептидных мостиков с помощью крупной субъединицы. У модельного организма Saccharomyces cerevisiae, малая субъединица (40S) представлена 33 рибосомными белками и 18S рибосомной РНК (rRNA), тогда как крупная субъединица (60S) представлена 46 рибосомными белками и 3 rRNAs (25S, 5.8S и 5S rRNA). См. table 1 in ref.1.
Начиная с первой атомной структуры рибосомальных комплексов эукариот, выявлена структурная сложность этой молекулярной машины2-4. В то время как рибосомные каталитические центры - место декодирования в малой субъединице и peptidyl transferase centre (PTC) в крупной субъединице - и др. рибосомные функциональные модули и ключевые архитектурные свойства, такие как центральный псевдоузелок в малой субъединице и центральные протруберанцы, GTPase activating centre (GAC), P0 ножка и туннель выхода полипептида (PET) в крупной субъединице, эволюционно законсервированы, рибосомы эукариот содержат множество дополнительных РНК расширений и белков5,6 (Supplementary Figs 1,2).
Сборка и тем самым доступность рибосомных субъединиц эукариот интимно связаны в доступностью питательных веществ, стрессами и клеточным циклом (rev. 7). Приблизительно 200 не-рибосомных факторов, включая белки, белковые комплексы и small nucleolar ribonucleoproteins (snoRNPs), необходимы для сборки малой и крупной рибосомных субъединиц (summarized in tables 3 и 4 в ref.8). Созревание pre-rRNAs для обеих субъединиц нуждается в эндонуклеолитическом и экзонуклеолитическом расщеплении (Fig. 1). Самостоятельные стадии этого процесса происходят сначала в ядрышке, затем в ядре и, наконец, в цитоплазме. Мы использовали дрожжевую номенклатуру для рибосомных белков.
 Fig. 1: Pre-rRNA processing in yeast.
Fig. 1: Pre-rRNA processing in yeast.
Consecutive pre-ribosomal RNA (pre-rRNA) processing stages produce rRNA intermediates through endonucleolytic and exonucleolytic removal of internal transcribed spacers (ITSs) and external transcribed spacers (ETSs). Precursors of mature rRNAs destined for the small ribosomal subunit are shown in green and those for the large ribosomal subunit in pink. Processing and assembly begin in the nucleolus, continue in the nucleoplasm and are completed in the cytoplasm. Sites in pre-rRNAs at which endonucleases cleave (A0, A1, A2, A3and C2) or exonucleases halt (B1) are highlighted in red at the step at which they occur. Each precursor RNA is designated by its size (assayed by velocity sedimentation on sucrose gradients) and by the site processed to generate that intermediate (for example, 27SA2). Each of these processing steps occurs within the indicated pre-ribosomal ribonucleoprotein particle. In rapidly growing yeast cells, processing and assembly occur mostly co-transcriptionally, with cleavage at the A0, A1 and A2 sites occurring in nascent pre-rRNAs, indicated by production of the 20S pre-rRNA (shown on the left). By contrast, in slowly growing yeast cells, processing occurs post-transcriptionally, with processing of the full-length 35S pre-rRNA (top) by cleavage at the A3 site to generate the 23S pre-rRNA and then at the A0, A1 and A2 sites (dashed arrow on the right). SSU processome, small-subunit processome.
 Fig. 2: Assembly of the small ribosomal subunit. Fig. 2: Assembly of the small ribosomal subunit.
Consecutive stages in the maturation of the small ribosomal subunit (40S) are shown, beginning with the earliest co-transcriptional steps in the nucleolus, the formation of the small-subunit (SSU) processome, nuclear export and final assembly in the cytoplasm. Small-subunit-specific portions of ribosomal DNA (rDNA) are depicted with colour-coding of the 5? external transcribed spacer (5' ETS); the 5?, central, ? major and 3? minor domains of the 18S ribosomal RNA (rRNA); and the internal transcribed spacer 1 (ITS1). The pre-rRNA cleavage sites A0, A1, D and A2 are indicated. Three ribosome particle intermediates are shown: the 5? ETS particle, SSU processome and the pre-40S particle. Pre-rRNA intermediates present in each particle are indicated in square parentheses beneath each particle. There are likely additional assembly intermediates not yet identified. Sequential association and dissociation of assembly factors and complexes of assembly factors are shown. Assembly factors and complexes for which structures have been determined are depicted in cartoon form, whereas those for which no structures are known are indicated only with text. The 5' ETS particle was inferred from purification of complexes assembled on 3? truncated pre-rRNAs. The earliest assembly intermediate for which cryo-electron microscopy structures were obtained is the SSU processome. Endonucleolytic cleavage at the A0, A1 and A2 sites and major structural remodelling (not shown) result in the release of assembly factors and the 5? ETS particle from the SSU processome. The resulting pre-40S particle assembles in the nucleus with a set of export factors and is rapidly exported to the cytoplasm, where the pre-40S particles engage in functional proofreading by joining with mature 60S subunits. The last assembly factors are released and the D site is cleaved to generate mature subunits containing 18S rRNA. Proteins that joined the growing SSU processome at an earlier stage are shown as transparent to highlight new components (not transparent). The 'wiggling' signs highlight components that are flexible in isolation. NPC, nuclear pore complex; Pol I, RNA polymerase I; snoRNP, small nucleolar ribonucleoprotein. Adapted with permission from ref.50, Elsevier.
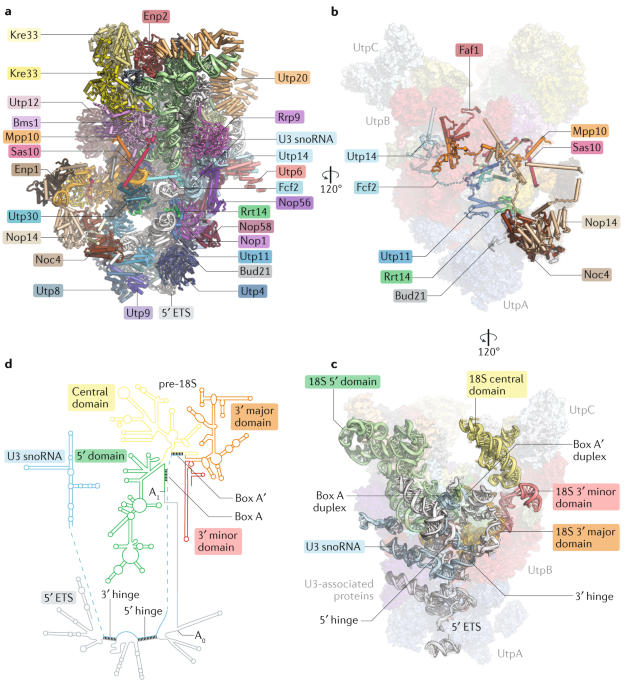 Fig. 3: Cryo-electron microscopy structure of a nucleolar small-subunit precursor.
Fig. 3: Cryo-electron microscopy structure of a nucleolar small-subunit precursor.
a-c | Three views of the small-subunit processome (PDB ID: 5WLC), highlighting the overall architecture (part a), organization of long peptide extensions that bridge distant regions in the particle (part b) and the interaction between pre-18S ribosomal RNA (rRNA), 5? external transcribed spacer (ETS) and U3 small nucleolar RNA (U3 snoRNA) (part c). d | Schematic representation of secondary structures of pre-18S rRNA and U3 snoRNA. The four domains of the pre-18S rRNA (5?, central, 3? major and 3? minor) are colour-coded. Base pairing of the U3 snoRNA 3? hinge and 5? hinge with the 5? ETS, and of the U3 snoRNA Box A and Box A? sequences with pre-18S rRNA are indicated. Protein complexes are outlined as transparent surfaces in parts b and c.
 Fig. 4: Assembly of the large ribosomal subunit. Fig. 4: Assembly of the large ribosomal subunit.
Consecutive stages in the maturation of the large ribosomal subunit (60S) are shown, from the earliest stages in the nucleolus, through stages in the nucleoplasm and finally in the cytoplasm. Large-subunit-specific portions of ribosomal DNA (rDNA) are depicted with colour-coding of the 5.8S ribosomal RNA (rRNA), the internal transcribed spacer 2 (ITS2), the 25S rRNA domains I-VI and the 3? external transcribed spacer (3? ETS). Six assembly intermediates for which cryo-electron microscopy (cryo-EM) structures have been determined are shown: state 1 or state A (state 1/A), state 2/B, state E, Nog2, Rix1-Mdn1 and Nmd3 particles. Pre-rRNA intermediates present in each particle are indicated in square brackets, and rRNA domains that have assembled into stable visible domains are depicted using the same colours of the rDNA. Note that some of the different particles contain the same pre-rRNAs but differ in structure and protein content (for example, state 1/A and state 2/B). There are likely additional assembly intermediates to be discovered. The association and dissociation of assembly factors is shown. Assembly factors for which structural information is available are depicted in cartoon form; those for which no structures are known are indicated with text only. The earliest pre-ribosomal particles present before state 1/A particles are formed co-transcriptionally and have not been visualized by electron microscopy. In the state 1/A and state 2/B particles, 25S rRNA domains I, II and VI and the 5.8S rRNA and ITS2 have begun to form and become stable, visible conformations. The transition from state 2/B to states C and D (which are not shown as particles), and then to state E, involves assembly of domains III, IV and V and includes early steps in the formation of the peptidyl transferase centre and polypeptide exit tunnel functional centres. Major structural remodelling occurs to form Nog2 particles, which translocate from the nucleolus to the nucleoplasm, where additional restructuring as well as quality control checkpoints are carried out to prepare particles for nuclear export. Upon entry into the cytoplasm, the remaining assembly factors are released, as the assembly and surveillance of functional centres is completed. The 'wiggling' signs highlight components that are flexible. NPC, nuclear pore complex.
Сборка рибосом в ядрышке характеризуется ко-транскрипционной ассоциацией факторов сборки рибосом с появляющейся pre-rRNA. Уменьшение конформационной свободы синтезированной pre-rRNA имеет целью формирование субдоменов как малой рибосомной субъединицы (Fig. 3), так и большой рибосомной субъединицы (Fig. 5). Во время сборки рибосом в ядре, относительные ориентации этих субдоменов уже ближе к зрелым конформациям в малой и большой субъединицах, но субдомены всё ещё подвергаются обширному ремоделированию (Fig. 6). Наконец, в цитоплазме происходит окончательная настройка и используется ступень контроля качества для тестирования функциональности обеих субъединиц их готовности к синтезу белка. Факторы сборки рибосом, ассоциированные с предшественниками малой рибосомной субъединицы хронологически представлены в Supplementary Table 1, а ассоциированные с большой рибосомальной субъединицей представлены в Supplementary Table 2.
 Fig. 5: Cryo-electron microscopy structures of large-subunit precursors. Fig. 5: Cryo-electron microscopy structures of large-subunit precursors.
a | Architecture of an early nucleolar pre-60S particle known as state 2 or state B (state 2/B; PDB ID: 6C0F), which includes the folded domains I, II and VI of pre-25S ribosomal RNA (rRNA) and the pre-5.8S rRNA. b | Architecture of a nucleolar pre-60S particle known as state E (PDB ID: 6ELZ, EMD 3891 and manual building). Note that domains III and V, and expansion segment 27 (ES27) of domain IV of the pre-25S rRNA, are now folded and visible. c | Architecture of the late nucleolar Nog2 particle (PDB ID: 3JCT). Here, domain IV is folded and visible with the 5S rRNA. Components already present in the previous particle are shown as transparent. d-f | Diagrams of the large-subunit rRNA secondary structure, showing the successive folding and stabilization of subdomains. Parts d-f depict rRNA organization of state 2/B, state E and Nog2 particles, respectively. Stably ordered RNAs (solid lines), largely ordered RNAs (dashed lines) and disordered RNAs (in light grey) are indicated with the same colour-coding as in parts a-c. CTD, carboxy-terminal domain; ITS2, internal transcribed spacer 2; NTD, amino-terminal domain.
 Fig. 6: Structural changes occurring in nucleolar and nuclear ribosomal precursors. Fig. 6: Structural changes occurring in nucleolar and nuclear ribosomal precursors.
a,b | Rearrangement of the relative orientations of pre-18S rRNA domains during the transition from the small-subunit processome (SSU processome; PDB ID: 5WLC) to the cytoplasmic pre-40S particle (PDB ID: 6FAI). The 5?, central, 3? major and 3? minor domains and the U3 small nucleolar RNA (U3) are indicated. These conformational changes are indicated by arrows in part a. c-e | Maturation of large-subunit particles near the Nog1 binding site in the nucleolar pre-60S particle (also known as state E; PDB ID: 6ELZ, manual building), the nucleolar Nog2 particle (PDB ID: 3JCT) and the nuclear Rix1-Mdn1 particle (PDB ID: 5JCS, manual building). In the transition from state E to Nog2 particles, the release of assembly factors Spb1, Noc3, Nip7 and Nop2 enables maturation of the polypeptide exit tunnel and stable docking of the 5S ribonucleoprotein (RNP) through Rpf2 and Rrs1. Subsequent formation of the nucleoplasmic Rix1-Mdn1 particle involves release of Rpf2 and Rrs1 to destabilize the pre-rotated state of the central protuberance and assembly of Sda1, the Ipi1?Rix1?Ipi3 complex and Mdn1 to stabilize the rotated state of the central protuberance. CTD, carboxy-terminal domain; NTD, amino-terminal domain.
В этом обзоре мы обсуждаем возникновение биохимических и структурных общих представлений при ядрышковой, ядерной и цитоплазматической сборке рибосомных субъединиц у S. cerevisiae. Основное внимание уделено ядрышковой стадии, к 4 ключевым понятиям сборки рибосом: планомерное уменьшение конформационной свободы pre-rRNA во время ранней ядрышковой стадии; хронология сборки факторов связывания, которая понуждается с помощью молекулярной мимикрии и молекулярных переключений, чтобы предотвратить состояние преждевременного образования складок или ступень процессинга и делает возможным своевременный прогресс в сборке; необратимые ключевые КПП (checkpoints), которые зависят от потребления энергии и преобразующих РНК энзимов, которые могут вызывать структурные изменения; и важность структурного и функционального считывания с функциональных центров обеих рибосомных субъединиц.
Nucleolar assembly
Почти у всех эукариот последовательность rRNA малой субъединицы расположена в тесной близости к 5' концу, а последовательности rRNA крупной субъединицы расположены в тесной близости к 3' концу rRNA предшественников. У S. cerevisiae, RNA polymerase I (Pol I) транскрибирует 35S rRNA предшественник, который помимо наружного и внутреннего транскрибируемых спейсеров - 5' external transcribed spacer (5' ETS), 3' ETS, internal transcribed spacer 1 (ITS1) и ITS2 - содержит 18S rRNA для малой субъединицы и 25S и 5.8S rRNAs для крупной субъединицы (Fig. 1). Pol III отдельно транскрибирует 5S rRNA, которая позднее интегрируется в крупную субъединицу.
Co-transcriptional pre-rRNA processing
Недавние структурные исследования показали, как транскрипция катализируется с помощью Pol I, которая является комплексом из 14 субъединиц, который транскрибирует rRNA с помощью соотв. набора факторов инициации9-14. Хотя известно, что или Pol I или Pol II могут быть использованы для транскрипции rRNA15, Pol I инактивируется посредством димеризации после истощения глюкозы, тем самым осуществляется прямая связь между доступностью пищевых компонентов и синтезом rRNA16.
Преобразование pre-rRNA может происходить или одновременно с транскрипцией или после транскрипции (rev. 17). Ранние доказательства пост-транскрипционного процессинга были получены в экспериментах, показавших, что виды 35S pre-rRNA могут синтезироваться первыми и что целый предшественник для обеих рибосомных субъединиц (далее обозначаемый как 37S rRNA) присутствует в 90S частицах18,19. Последующие работы показали, что преобразования во время транскрипции pre-rRNA могут быть видимы на Miller spreads20 и что в быстро растущих клетках, большая часть процессинга (~70%) происходит одновременно с транскрипцией21.
При совпадении с транскрипцией rRNA подвергается ковалентным модификациям, большинство из которых концентрируется в функционально важных доменах и, как полагают, тонко контролируют структуру и функцию rRNA22. Разные классы факторов ассоциированы с модификациями pre-rRNA. Среди RNA-содержащих классов, те, что включают snoRNPs, которые могут катализировать или pseudouridylation (H/ACA snoRNPs) или 2'-O-ribose methylation (box C/D snoRNPs) pre-rRNA. H/ACA snoRNPs и box C/D snoRNPs наводятся с помощью snoRNAs, которые содержат последовательность элементов, которые образуют пары оснований с rRNA мишенью, а структурные мотивы для связывания обеспечиваются белковыми кофакторами. Box C/D-ассоциированные белки включают methyltransferase Nop1, гетеродимер Nop56-Nop58 и Snu13; H/ACA-ассоциированные белки, включая pseudouridine synthase Cbf5 и Gar1, Nop10 и Nhp2 (refs23,24).
Двумя др. snoRNPs, которые выполняют центральную роль в сборке рибосом эукариот, являются U3 snoRNP и RNase MRP (mitochondrial RNA processing). Семейство box C/D U3 snoRNP является ключевым структурным организатором для сборки малой субъединицы и для одновременного с транскрипцией расщепления pre-rRNA для малой субъединицы по сайтам A0, A1 и A2 (Fig. 3c,d), но это не является результатом метилирования 2'-O-ribose. Напротив, RNase MRP катализирует расщепление по сайту A3 (refs25,26), сайту, который также ассоциирует с пост-транскрипционным расщеплением (Fig. 1).
Помимо RNA-обеспечиваемых модификаций РНК, 19 RNA helicases, включая DEAD-box и DEAH-box геликазы, участвуют в сборке рибосом у дрожжей. 7 из этих факторов (Dbp4, Dbp8, Dhr1, Dhr2, Fal1, Rok1 и Rrp3) участвуют в сборке малой субъединицы, 9 геликаз (Dbp2, Dbp3, Dbp6, Dbp7, Dbp9, Dbp10, Drs1, Mak5 и Spb4) участвуют в сборке крупной субъединицы, а три (Has1, Mtr4 и Prp43) участвуют в сборке обеих субъединиц (rev. 27,28). Кроме того, потребляющие энергию, энзимы, включают GTPases , такие как Bms1, Nog1, Nog2, Nug1, Lsg1 и Efl1; ATPases, такие как Rio1, Rio2 и Fap7; и AAA-ATPases (Mdn1, Drg1 and Rix7) (rev. 29). Роль этих факторов заключается в управлении сборкой рибосом косвенным образом. Многие из факторов сборки обладают непредсказуемыми ферментативными функциями, но содержат РНК-связывающие мотивы и могут наблюдаться в структурах по связыванию rRNA. Наконец, сборка ряда рибосомных белков в промежуточные рибосомы осуществляется с помощью соотв. шаперонов, которые делают возможным их укладку во время трансляции, экскортируя их с ядро или помогая вставлять их в pre-ribosomal частицы, находящиеся в процессе сборки (rev. 30,31).
Связь между окончательными структурами, наблюдаемая в Miller, и ассоциация pre-rRNA с факторами сборки малой субъединицы рибосом оставались длительное время неизвестными. Окончательная шарообразная структура, присутствующая при Miller spreads первоначально считались как содержащие pre-rRNA 32, но более поздние исследования идентифицировали их как комплексы для преобразования rRNA, которые содержат 5' ETS у Xenopus laevis33 и нуждаются в U3 snoRNA 34. Идентификация small-subunit processome (SSU processome), как U3 snoRNA-ассоциированных пре-рибосомных частиц, содержащих большинство факторов сборки рибосом, предоставило первые доказательства связи ранее наблюдаемых структур с определенным макромолекулярным комплексом 35. В др. исследовании сходные частицы содержали малую субъединицу, но были лишены крупной субъединицы, были описаны факторы биогенеза, которые были названы 90S pre-ribosome, т.к. было предположено, что они содержат 35S pre-rRNA 36. Т.к. все современные данные подтверждают, что обе частицы в самом деле являются SSU processome (т.к. они не содержат 35S pre-rRNA и лишены факторов сборки крупной субъединицы) 37-40, поэтому мы обозначили эти частицы как SSU processome.
Nucleolar assembly of the small subunit
Как ранняя стадия сборка ядрышковых рибосом происходит одновременно с транскрипцией, изучение временных ассоциаций с pre-ribosomal частицами показало, что большинство факторов сборки представляют основное узкое место, поскольку этот процесс происходит динамически со 100-200 ribosomal DNA (rDNA) локусами. Использование дрожжевых pre-rRNA воспроизводит мечение с помощью RNA aptamers, что позволяет изучать эти ранние ступени сборки малой субъединицы рибосом41,42 и крупной субъединицы43 в связи с транскрипцией. Вместе с характеристикой белковых комплексов44-47 и исследований по истощению48,49, эти данные предоставляют нам современную модель одновременной с транскрипцией сборки малой рибосомной субъединицы (rev. 50) (Fig. 2).
Во время формирования SSU processome, pre-rRNA подвергается химическим модификациям и образуются 4 домена 18S rRNA (5', central, 3' major and 3' minor домена). Эти субдомены содержат независимые вторичные структурные элементы, которые в зрелой малой субъединице формируют 3D структуру 18S rRNA (Supplementary Figure 2). Важно, что РНК и белковые факторы участвуют в уменьшении конформационной свободы pre-rRNA. Это двойное участие используется. чтобы ориентировать 4 субдомена в конформации, которая предотвращает их преждевременное складывание, в тоже время позволяет каждому домену собираться отдельно в условиях энкапсуляции. Эта энкапсуляция является концептуально отличающейся от созревания крупной субъединицы (discussed below), которая происходит модулярным способом, когда субдомены связываются со многими изолированными факторами сборки.
Формирование 5' ETS pre-ribosomal частиц 41 создает первый каркас, на котором располагаются 5' ETS; белковые комплексы UtpA, UtpB и Mpp10; U3 snoRNP (важный RNA chaperone комплекс); и мелкие индивидуальные белки и создают архитектурную поддержку для субдоменов SSU, которые образуются последовательно41,42 (Fig. 3a,b).
U3 snoRNA выполняет центральную роль в формировании SSU processome, т.к. её основания спариваются с и тем самым делают более жестким регионы обоих 5' ETS (с их 5' and 3' hinges) и pre-18S РНК (с их Box A и Box A')39,50-56 (Fig. 3c,d). Тесная близость этих сайтов связывания внутри 5' региона U3 snoRNA создает критическое пространственное ограничение, которое предопределяет топологию зрелых частиц. Сходным образом, некоторые ранние мультимодальные связывающие белки (Utp11, Sas10, Mpp10 и Fcf2) ограничивают pre-rRNA домены внутри частиц путем соединения или с белком или с РНК элементами57,58.
Кроме того, многие факторы выполняют временные роли в биогенезе ранних частиц малых субъединиц и могут присутствовать во время транскрипци только перед дроблением в сайте A2. Они включают как малые РНК, такие как U14, snR10 и snR30 (refs59-61), так и белки, которые ассоциируют с каждым из субдоменов 18S rRNA62-64. По сравнению с более стабильно ассоциированными факторами сборки, которые формируют часть зрелых SSU processome, роль этих преходящая и исследована слабо (Fig. 2).
События расщепления РНК представляют собой необратимые ступени, которые отделяют pre-rRNAs др от др. и от ITSs или ETSs. Во время одновременного с транскрипцией процессинга pre-rRNA расщепление происходит в сайтах A0, A1 и A2, тем самым высвобождается предшественник 20S pre-rRNA21; напротив, во время пост-транскрипционного процессинга, RNase MRP расщепляет по сайту A3, чтобы сгенерировать предшественник 23S, который преобразуется ещё дальше25,26 (Fig. 1). Функция 23S rRNA ясна нецеликом, т.к. она также ассоциирована с аберрантным процессингом pre-rRNA в ответ на истощение факторов сборки малой субъединицы и рибосомных белков крупной субъединицы65,66. Тогда как недавние доказательства подтверждают, что Utp24 является нуклеазой, ответственной за расщепление по сайтам A1 и A2, нуклеаза, ответственная за расщепление по сайту A0 пока не установлена67,68.
Недавно были определены с помощью cryo-EM структуры SSU processome, которые проиллюстрировали её архитектуру37-39 и почти атомного уровня структуры56,57. Последнее вместе с данными кристаллографии 4,69-77 и данными межбелковых взаимодействий78-80 предоставили молекулярный образ (snapshot) самого раннего стабильного предшественника малой рибосомной субъединицы. Из-за очень высокого сходства частиц, полученных при использовании трех разных экспериментальных условий (клетки во время экспоненциального роста56, голодающие клетки57 и истощенные по Mtr4 клетки39), очень вероятно, что SSU processome являются промежуточными, которые формируются при физиологических условиях.
Дрожжевые SSU processome предоставляют заключенные в капсулу микроусловия для субдоменов rRNA малой субъединицы. Основание частицы формируется вокруг 5' ETS и белковых комплексов UtpA и UtpB, которые эволюционно связаны и действуют как молекулярные шапероны в основании и боках частиц, соотв. (Fig. 3). Верхушка частицы содержит 5', центральный и 3' домены rRNA, каждый из которых помещен в отдельный регион SSU протеосомы. Взаимосвязь большинства белков, содержащих длинные пептидные расширения, таких как Mpp10, Utp11 и Sas10 (Fig. 3b), и спаривание оснований U3 snoRNA с 5' ETS и 18S rRNA (Fig. 3c,d) предоставляют дальнейшее структурное подтверждение для частиц. Факторы сборки рибосом, которые стабильно ассоциируют с субдоменом pre-18S rRNA преимущественно действуют как локальные стабилизаторы элементов РНК57.
Как helical-repeat-содержащие белки (Nop14, Noc4, Rrp5, Utp10 и Utp20), так и некоторые ферменты, такие как methyltransferase Emg1 (ref.75), acetyltransferase-helicase Kre33 (ref.81) и GTPase Bms1 (refs82,83), расположены на наружных регионах SSU processome. В то время как роль helical-repeat-содержащих белков является чисто структурной, временную же последовательность действия ферментов на энкапсулированные pre-18S rRNA ещё предстоит определить.
Ключевая структурная роль SSU processome заключается в обеспечении (chaperoning) сборки каждого субдомена 18S rRNA, т.е. в предупреждении преждевременного образования центрального псевдоузла (pseudoknot)57. Однако, во всех доступных на сегодня структурах, SSU processome содержат крайне запутанные pre-18S rRNA, которые д. быть высвобождены из SSU processome для дальнейшего созревания (Fig. 2). Чтобы достичь этого, РНК геликазы. таке как Dhr1, которые участвуют в раскручивании U3 snoRNA84,85, необходимы для освобождения 18S предшественника от U3 snoRNA. Сходным образом, расщепление по сайтам A1 и A2 с помощью Utp24 необходимо, но невозможно в доступных структурах, поскольку активный сайт Utp24 недоступен57.
Наконец, процессинг SSU processome, в особенности удаление 5' ETS, нуждается в экзосомах и белках, взаимодействующих с экзосомами, таких как Utp18, Sas10 и Lcp5 (refs57,86,87).
Assembly of the large ribosomal subunit
Структура крупной субъединицы более тщательно разработана; её сборка следует по более сложному пути. 25S rRNA крупной субъединицы состоит из 6 законсервированных доменов вторичной и третичной структуры, а именно (5' -> 3') домены I-VI, которые ещё больше спутаны др. с др., чем домены 18S rRNA в малой субъединице (Supplementary Figure 2). Обращенная к растворителю поверхность крупной субъединицы включает домены I и II и 5.8S rRNA, тогда как интерфейс между субъединицами содержит функциональные центры, которые включают домены IV и V. Домены III и IV образуют мостики между краем solvent-exposed поверхности и интерфейсом субъединиц; домен III также связывает РНК домены в основании субъединицы. 5.8S rRNA расположена между доменами I и III, а 5S rRNA находится на верхушке доменов II и V (Supplementary Figure 2).
Поскольку мы лишены cryo-EM структур для более ранних стадий сборки 60S субъединицы в ядрышке, то наше понимание базируется только на биохимических и генетических исследованиях процессинга pre-rRNA и факторов сборки, которые участвуют в инициальной упаковке pre-60S ribosomal ribonucleoproteins (rRNPs). Напротив, cryo-EM изображения ряда промежуточных сборок, следующих за самой ранней ядрышковой стадией сборки, предоставляют более детальные модели более поздних стадий биогенеза крупных субъединиц (Figs 4, 5). Сборка крупной субъединицы начинается вместе с транскрипцией, при этом возникают ковалентные модификации pre-rRNA, под руководством snoRNPs. В то же самое время происходят укладка длинной, гибкой pre-rRNA в более компактную и стабильную конформацию и расщепление и процессинг ITS1 и 3' ETS (Fig. 1). К растворителю обращенная поверхность крупной субъединицы формируется первой путем укладки 5.8S rRNA вместе с 25S доменами I и II (seen in state 1 or state A (state 1/A)) and then with domain VI (state 2/B) into stable structures (Fig. 5a,d). Вмешательство доменов III, IV и V первоначально гибкое и связано с факторами сборки. Т.к. 25S rRNA домены III, IV и V становятся структурно видимыми как и соединение уже уложенных в складки доменов I, II и VI (Fig. 5b,e), то инициальные стадии конструкции функциональных центров крупных субъединиц (PTC и PET) становятся очевидными (state E). Во время следующего перехода от ядрышка к нуклеоплазме (Figs 4; 5c,f), обмены многочисленных белков делают возможным др. крупное событие ремоделирования, приводящее к экспорту из ядра. Затем в цитоплазме, pre-60S рибосомы подвергаются финальной стадии созревания, включая удаление оставшихся факторов сборки, последних немногих рибосомных белков сборки и тестирование функциональности центров.
Nucleolar stages of large-subunit assembly
Среди факторов сборки, участвующих в самой ранней одновременной с транскрипцией стадии сборки крупной субъединицы имеются несколько белков, которые содержат множественные РНК-связывающие домены или α-helical повторы, такие как Rrp5, Mak21, Noc2 и Nop4. Эти белки могут оказаться способны к компакции rRNA путем формирования ригидных каркасов, чтобы направлять и стабилизировать упаковку в складки РНК88-95. В Miller spreads, окончательные шарообразные структуры, соответствующие пре-рибосомным частицам34, имели более рыхлую структуру у мутантов, лишенных этих факторов сборки92. Факторы ранней сборки Npa1, Npa2, Rsa3 и Nop8 иРНК геликаза Dbp6 образуют стабильный комплекс, который также может выполнять структурную функцию 96-98. 6 др. РНК геликаз (Dbp2, Dbp3, Dbp7, Dbp9, Mak5 и Prp43) также необходимы для этой очень ранней сборки, скорее всего. чтобы ремоделировать РНК или RNPs (rev. 28,60,99). Однако, сайты связывания и точные мишени для этих энзимов пока неизвестны.
Интересно, что расщепление в сайтах A2 и A3 в ITS1 сцеплено с транскрипцией или процессингом последовательностей в несколько kilobases вдали от этих сайтов. Одновременное с транскрипцией расщепление в A2 сайте происходит только когда Pol I оказывается ~1.2-1.5 kb ниже его, в домене I и домене II части последовательности 25S rDNA21,100, а расщепление в сайте A3 (вблизи места. которое станет 5' концом 27SB pre-rRNA) происходит только после завершения транскрипции и процессинга 3' ETS на несколько kilobases ниже101. В точности, как эти расщепления связаны с нижестоящими событиями, пока неизвестно, но крупномасштабная, как и локальная упаковка РНК, обеспечиваемая с помощью белка связывания, может быть необходима для создания субстрата для расщепления. Напр., связывание Rrp5 вблизи сайта процессинга pre-rRNA как в SSU processome (A2 site)41,42,57, так и pre-60S частицах (A3 site) может регулировать время расщепления в сайтах и координировать сборку обеих субъединиц88,91,92,102. Половина N-терминального домена Rrp5, которая содержит множественные РНК-связывающие мотивы, которые связывают вблизи сайта расщепления A3 в ITS1, необходима для A3 расщепления. С-терминальная половина Rrp5, содержащая дополнительные РНК-связывающие мотивы, связывает поперечно вблизи сайта расщепления A2 в ITS1 и необходима для A2 расщепления. Т.о., соотв. упаковка в складки последовательности ITS1, также как и домена I и домена II, связывающая N-терминальный домен Rrp5, может быть необходима для передачи сигнала об успешной инициации сборки крупной субъединицы прежде, чем SSU processome может быть отделана от pre-60S частиц с помощью расщепления по сайту A2. Сходным образом, 3D близость 5' конца и 3' конца 27SB pre-rRNA подтверждает, что их процессинг может быть скоординирован, вообще-то посредством межбелковых и белок-РНК взаимодействий Rrp5 и рибосомного белка L3, который связан с этими концами66,92.
Cryo-EM структуры103-105 начинают демонстрировать, как домены rRNA ранних ядрышковых субъединиц pre-60S удерживаются вместе: факторы сборки и рибосомные белки формируют мостики внутри и между каждым доменом rRNA. Напр., в состоянии 2/B частиц, три фактора сборки (Nop15, Cic1 и Rlp7) соединяются с ITS2 и 5 др.(Nop7, the Erb1-Ytm1 complex, the RNA helicase Has1 и Nop16) собираются в домене I, окружающем ITS2 (Fig. 5a,d). Эти 8 факторов вместе в некоторыми рибосомными белками, связывающимися поблизости, могут помочь стабилизировать каждую очень раннюю складку, осуществляемую с помощью спаривания оснований 5.8S rRNA с последовательностями в домене I (Supplementary Figure 2). 9 др. факторов сборки (Mak16, Rpf1, Nsa1, Rrp1, Ssf1 в комплексе с Rrp15, Rrp14 и Ebp2 в комплексе с Brx1) образуют кольцо вокруг центра частицы в состоянии 2/B, чтобы связать мостиками домены I, II и VI др. с др.103 (Fig. 5a). 4 др. фактора сборки (GTPase Nog1, eIF6, Rlp24 и Mak11) соединяются с доменом VI вблизи интерфейса с доменом V (Fig. 5a).
Хотя большинство рибосомных белков в rRNA домене I и домене II уже находятся в своей зрелой конформации в ядрышковых частицах, сборка этих доменов заканчивается только, когда остальные факторы сборки, с ними связанные, высвободятся из пре-рибосомных частиц. Напр., факторы сборки, такие как Ebp2-Brx1 приспособлены, чтобы предупреждать от преждевременного образования RNA-RNA взаимодействий между доменами I и V (Fig. 5a) и должны быть удалены, чтобы сделать возможной нижестоящую ступень созревания103. Такой переход прекрасно иллюстрирует важный принцип сборки рибосом - избавление от факторов сборки из pre-ribosomal частиц также важно, как их поступление или присутствие.
В ядрышке функциональные центры крупной субъединицы (GAC, PTC и PET) начинают проявляться во время перехода от состояния 2/B частиц к состоянию C, D и E частиц104 (Fig. 5a,b,d,e). Напр., факторы сборки Nop2, Nip7, Noc3 и methyltransferase Spb1 соединяются с rRNA доменами IV и V с целью формирования PTC104 (Fig. 5b). Сборка ободка (rim) вокруг выхода из PET заканчивается путем освобождения факторов сборки Ssf1-Rrp15 и Rrp14 и с помощью стабильной ассоциации рибосомных белков L19, L25 и L31 (Fig. 4). Ssf1 располагается в той же самой позиции в ядрышковых промежуточных образованиях (Fig. 5a) , что и L31 в зрелых рибосомах103. Т.о., удаление Ssf1 вместе с Rrp15 и Rrp14 может позволить дальнейшую ассоциацию L31, L19 и L25 , чтобы завершить образование PET ободка.
Во время перехода от состояния D к состоянию E, происходит образование преддверия (vestibule) - наружной, широкой порции PET - это становится возможным при выходе из ансамбля фактора Rpf1. N-терминальная часть Rpf1 располагается в том месте, где будет преддверие, которое образуется как только Rpf1 будет удален 103-105. Демонтирование Rpf1 из ранних pre-rRNPs происходит вместе с соседними белками Mak16, Rrp1 and Nsa1 (Fig. 4). Скорее всего, освобождение от этих факторов сборки делает возможным прямое удаление Nsa1 с помощью AAA-ATPase Rix7 (ref.106).
Considerable remodelling upon transit of pre-60S ribosomes from the nucleolus to the nucleoplasm
По крайней мере, 9 факторов сборки уходят и 11 факторов проникают в pre-60S частицы во время их перехода из ядрышка в ядро. Это доказывается различиями между состоянием E частиц и Nog2 или Rix1-Mdn1 частиц104,107-109 (Figs 4; 5b,c,e,f; 6c-e). Эти и описанные выше обмены белков запускают многочисленные нижестоящие события, включая дальнейшую конструкцию функциональных центров, таких как PTC и PET, также как и стабильное закрепление ранее гибких центральных протруберанцев и расщепление ITS2, чтобы инициировать их удаление.
PET подвергается дополнительным ступеням конструкции во время поздней ядрышковой стадии сборки крупных субъединиц, когда Ebp2, Brx1, Noc3, Spb1, Nop2 и Nip7 выходят, а Nog2 и Rsa4 соединяются с доменом V (Figs 5b,c; 6c,d). Сравнение ядрышкового состояния E частиц и Nog2 показывает, что в состоянии E частиц факторы сборки Spb1, Ebp2 и Noc3 блокируют формирование зрелой структуры rRNA спирали в доменах II и V, которые образуют порцию стенки РНК зрелого PET. Т.о., освобождение от этих факторов во время перехода от ядрышковых частиц в состоянии E к частицам Nog2 может сделать возможным формирование функционального PET. Окружение из Nog1 также становится предметом крупного ремоделирования во время перехода от ядрышковых к pre-60S частицам (Fig. 6c) через поздние ядрышковые Nog2 частицы (Fig. 6d) к ядерным Rix1-Mdn1-содержащим pre-60S частицам (Fig. 6e). Особенно примечательным является инсерция С-терминального расширения Nog1 в PET, почти назад через PTC108. Это может сделать возможным продолжение сборки туннеля, т.к. во время этого интервала спирали, которые формируют стенку туннеля, переходят от незрелого, неупакованного состояния к зрелой конформации. Альтернативно или в дополнение расширение Nog1 может контролировать собственно сборку туннеля или предупреждать проникновение др. молекул.
Центральные протурберанцы, поверх зрелой 60S субъединицы, включают 5S RNP (рибосомные белки L5 и L11, связанные с 5S rRNA) и домен V спиралей 80 и 82-88 (refs3,4). Поскольку 5S rRNA транскрибируется с разных генов с др. трех rRNAs, она должна собираться отдельно в пре-рибосомы. Соотв. шаперон Syo1 облегчает сборку 5S rRNA с L5 и L11 в цитоплазме и нуклеоплазме, а факторы сборки Rpf2 и Rrs1 необходимы для ассоциации этого 5S rRNP с пре-рибосомными частицами110-112. Хотя 5S RNP ассоциирует с более ранними pre-60S частицами, по-видимому, соединяясь со спиралью центральных протруберанцев, она не видна при cryo-EM вплоть до формирования Nog2 частиц, скорее всего, благодаря гибкости незакрепленных центральных протруберанцев в ранних сборках промежуточных образований108. Благодаря сайтам связывания Nip7 и Nop2 в ранних ядрышковых частицах существуют почти рядом или перекрываются сайты Rpf2 и Rrs1 в поздних Nog2 частицах104, переход 5S rRNP в более стабильную конформацию в пре-рибосомах может зависеть частично от удаления комплекса Nop2-Nip7 из пре-рибосомных частиц перед формированием Nog2 частиц (Figs 5b,c; 6c,d). Rpf2, Rrs1, L5 и L11 необходимы также для сборки Nog2 с доменом V rRNA113, подтверждая, что стабилтное закрепление центральных протруберанцев может влиять на дальнейшую конструкцию PTC в домене V.
Расщепление ITS2 связано с формированием PET и PTC и с закреплением центрального протруберанца. Первоначальный запуск этого расщепления, по-видимому, высвобождает соседние факторы сборки109,114-118. AAA-ATPase Mdn1 соединяется с фактором сборки Ytm1, и высвобождает Ytm1 и ассоциированный белок Erb1, N-терминальное расширение которого располагается вокруг pre-25S rRNA домена I проксимальнее ITS2 (refs103,104). Факторы сборки, которые взаимодействуют с этим длинным расширением из Erb1, также уходят в это время и их выход преимущественно связан с высвобождением Erb1. Хотя мы не знаем, как эндонуклеаза Las1 специфически воздействует на пре-рибосомные частицы, чтобы отщеплять ITS2, мутации в рибосомных белках или факторах сборки (включая Mdn1, которые предупреждают высвобождение этих белков, которые находятся проксимальнее ITS2, также блокируют его расщепление с помощью Las1. Это наблюдение подтверждает, что ремоделирование в rRNA домена I вблизи ITS2 в пре-рибосомных частицах необходимо для создания сосбственно субстрата для Las1 (refs109,113,114,118,119).
Сравнение с Las1, делает яснее, как экзосома рекрутируется в пре-рибосомные частицы, чтобы преобразовать ITS2, как только он будет расщеплен 120. Используемый механизм, который комбинирует молекулярный сдвиг и молекулярную мимикрию, способен ремоделировать домен вокруг ITS2. Mtr4, который является компонентом геликазы экзосомы соединяется с адапторным белком Nop53 в пре-рибосомных частицах86,121. Поскольку Erb1 и Nop53 используют одни и те же мотивы, чтобы связать фактор сборки Nop7 в пре-рибосомных частицах86,103,121, удаление Erb1 необходимо для связывания Nop53 и тем самым для привлечение экзосомы.
Интересно, что хотя расщепление C2 сайта в ITS2 сцеплено с более ранними событиями ремоделирования, неспособность расщепления или преобразования ITS2 , по-видимому, не затрагивает нижестоящие ступени сборки 60S субъединицы вплоть до поздних цитоплазматических стадий 109,122,123. Вследствие истощения или инактивации Las1 или Mtr4, не происходит удаления ITS2. Однако, созревание субъединицы продолжается, хотя возникающие в результате рибосомы сохраняют факторы Nop15, Cic1 и Rlp7, связанные с ITS2. ITS2-содержащие 60S субъединицы каким то образом обходят механизмы ядерного надзора и экспортируются в цитоплазму, но оказываются дефектными в отношении трансляции и подвергаются целенаправленному обороту с помощью комплекса контроля качества рибосом и цитоплазматических экзосом 123.
Nucleoplasmic stages of large-subunit assembly
По достижению нуклеоплазмы pre-60S частицы подвергаются дополнительному ремоделированию, т.к. они становятся компетентными к экспорту в цитоплазму. Сюда входит удаление ITS2 и реконструкция центрального протруберанца, а также структурное считывание функциональных центров.
ITS2 удаляется последовательно с помощью трех комплексов нуклеаз: Las1-Grc3-Rat1-Rai1 комплекса, экзосомы и Ngl2-Rex1-3 (refs17,86,121,124-130). Эндонуклеаза Las1 расщепляет C2 сайт в ITS2,чтобы сгенерировать 7S и 25.5S pre-rRNAs (Fig. 1). Укорочение 25.5S pre-rRNA до 25S rRNA осуществляется с помощью киназы Grc3 и завершается с помощью нуклеаз Rat1 и Rai1. Экзосома из 14-субъединиц120 удаляет с 3' конца 7S pre-rRNA все, кроме наиболее 5' 30 нуклеотидов из ITS2 и затем на второ2й ступени все кроме последних 8 нуклеотидов из 6S pre-rRNA130. В цитоплазме Ngl2 и Rex1-3 катализируют процессинг 6S предшественника в зрелую 5.8S rRNA131.
Формирование структуры зрелого центрального протруберанца нуждается в широко-масштабной структурной перестройке, которая служит в качестве КПП (checkpoint) в нуклеоплазму перед экспортом в ядро. 5S RNP первоначально помещается 'обратно' на верхушку pre-60S частиц, затем поворачивается ~180°, чтобы принять свою зрелую конфигурацию. В то же самое время сопровождающие центральный протруберанец спирали 80 и 82-88, также подвергаются крупно-масштабной структурной перестройке. Эти события ремоделирования используют альтерации многочисленных белок-РНК контактов, пока неясно, что заставляет их оставаться108,132. Непосредственно перед ротацией центрального протруберанца, Rpf2 и Rrs1 устраняются. Эти два фактора сборки связывают 5S RNP со спиралями центрального протруберанца в rRNA домене V на вершине pre-60S рибосомы108,133-135. Т.о., их выход может запускать дестабилизацию состояния перед ротацией. Устранение Rpf2 и Rrs1 может быть вызвано соединением фактора сборки Sda1 с пре-рибосомными частицами и с помощью его конкуренции с Rpf2 для частичного перекрывания сайтов связывания (Fig. 6d,e). После ротации Rix1-Ipi1-Ipi3 комплекс и AAA-ATPase Mdn1 устанавливают множественные контакты с пре-рибосомными частицами, чтобы стабилизировать состояние ротации119 (Fig. 6e). Гидролиз АТФ с помощью Mdn1 активирует Nog2 GTPase, приводя к её высвобождению, а также к высвобождению Sda1, Rix1-Ipi1-Ipi3, Mdn1 и Rsa4 (Fig. 4).
Чтобы минимизировать продукцию нефункциональных рибосом, дрожжи используют структурную проверку (proofreading) функциональных центров рибосом во время ступеней сборки в ядре, это связано с экспортом из ядра (rev.136). Фактор экспорта Arx1 и его партнер Alb1 собираются на ободке вокруг PET с помощью связывания рибосомных белков L19, L25, L26 и L35 (refs107,137). Т.о., перед экспортом пре-рибосомных частиц связывание Arx1 может служить проверкой собственно приспособленности этих рибосомных белков в выходе из туннеля, где ряд факторов шаперонов и мишеней целенаправленно воздействует на синтезируемые полипептиды во время синтеза белка 138,139. GTPase Nog2 (ref.140) и соседние GTPases Nog1 и Nug1 и РНК геликаза Dbp10 (refs108,141) могут влиять или или тщательно изучать собственно формирование PTC: после высвобождения Nog2, такое соседство РНК выглядит всё более похожим на зрелый PTC 142. Уход Nog2 делает возможным сборку важного фактора экспорта Nmd3, сайт связывания которого соответствует части из L1 ножки посредством E сайта и P сайта в направлении sarcin-ricin loop 140,142,143. Т.о., образование почти зрелого PTC может иметь значение для готовности к экспорту пре-рибосомных частиц. Созревание P0 ножки также связано с экспортом в ядро: фактор сборки Yvh1 позволяет высвобождать фактор сборки Mrt4 из незрелой ножки, делая возможным связывание факторов экспорта Mex 67 и Mtr2 (refs144,145).
Cytoplasmic stages of large-subunit assembly
Финальная стадии созревания крупной субъединицы происходит в цитоплазме, где оставшиеся факторы сборки диссоциируют с частиц пре-рибосом и обратно отправляются в ядро для повторного использования и , по крайней мере, 9 рибосомных белков инкорпорируются (rev. 136,146). Удаление каждого из этих белков управляется с помощью GTPase или ATPase и связано с вышестоящей и нижестоящей ступенями иерархического пути147. Важно, что каждый из функциональных центров крупной субъединицы ассоциирован с факторами сборки вплоть до поздней стадии и каждый функциональный центр подвергается функциональному тестированию.
Фактор сборки Rlp24 рекрутирует и активирует AAA-ATPase Drg1, которая катализирует замещение Rlp24 гомологичным рибосомным белком L24 (refs147-149). Поскольку L24 лишен мотива, присутствующего в Rlp24, который распознается с помощью Drg1, то это замещение является однонаправленным молекулярным переключением. Подавление Drg1 с помощью diazaborine блокирует высвобождение Rlp24 в цитоплазму, но также (прямо или косвенно) высвобождает соседствующие с Rlp24 факторы сборки Nog1, Nug1 и Nsa2 (refs148,150).
Факторы сборки Rei1 и Jjj1 и Hsp70 (chaperone)-подобная ATPase Ssa необходимы для высвобождения Arx1 и Alb1 из ободка вокруг выхода из PET151-154. Rei1 соединяется по соседству с Arx1 и внедряет свой С-терминальный домен в PET, продвигаясь почти назад в PTC137,154. После удаления Rei1 из PET, он замещается Reh1. Как удаление Arx1 и Rei1 связано с последующих уходом факторов сборки eIF6 и Nmd3 неизвестно, но это может быть связано с коммуникациями с PTC посредством 'хвоста' от Rei1, расположенного внутри PET.
Nmd3 и eIF6 высвобождаются на финальных ступенях биогенеза крупных субъединиц, вообще-то сочетанно с помощью сложных танцев факторов сборки143,147,155,156. Эти ремодельеры включают GTPases Lsg1 и Efl1, рибосомный рециклинг фактор-подобный белок Sdo1 и рибосомный белок L10. Nmd3 помогает сборке L10, тогда как сборка Efl1 на GAC, которая сопровождается ротацией внутри Sdo1, делает возможной высвобождение eIF6 с помощью Efl1. Домен P петли в L10 также необходим для высвобождения eIF6 с помощью Efl1. Lsg1 необходим для выхода Nmd3.
Т.о., структура и функция A сайта, P сайта, E сайта и GAC, по-видимому, проверяются с помощью Nmd3, Sdo1, L10 и Efl1, чтобы сделать возможными последние ступени сборки. Между тем PET может подвергаться последовательной проверке с помощью Nog1, Rei1 и Reh1 в ходе сборки. Успешное завершение этих цитоплазматических ступеней освобождения от факторов сборки связано с функциональными центрами 60S субъединиц, это делает возможной сборку рибосомных белков и лицензирование каждого домена на участие в белковом синтезе.
SSU assembly: nucleocytoplasmic stages /div>
3' домен в 18S rRNA, который позднее формирует головку зрелой малой субъединицы, становится предметом крупных ступеней ремоделирования между ядрышковыми SSU processome и цитоплазматическими pre-40S частицами57,157,158 (Fig. 6a,b). В то время как факторы, такие как Enp1, Pno1 и Rrp12 остаются ассоциированными с 3' доменом, все остальные факторы SSU processome отделяются от pre-40S субъединицы с помощью механизма, которые всё ещё плохо изучен. Экспорт в ядро предшественников малой субъединицы происходит RanGTP-зависимым и Crm1-зависимым способом (rev. 31). Кроме того, ядерные pre-40S частицы нуждаются в Rrp12 для эффективного экспорта в цитоплазму159, и Rrp12 отсутствует в цитоплазматических промежуточных образованиях у дрожжей, которые недавно были открыты157-160.
Во время перехода от ядрышка к цитоплазме pre-40S рибосомы также приобретают серию самостоятельных нуклеоцитоплазматических факторов сборки, включая Tsr1, Rio2, Dim1 и Ltv1. Ltv1 замещает пептид Nop14 на поверхности Enp1 (refs 39,57), а каталитически неактивный Tsr1 (ref.161) замещает структурно родственную GTPase Bms1 (refs82,83) вблизи 5' домена pre-40S частицы (Fig. 2). Др. важная ступень заключается в формировании напоминающей клюв структуры из малой субъединицы, которая в зрелой структуре формируется с помощью rpS31, rpS12, rpS10, rpS3 и rpS20. В поздних pre-40S частицах, Enp1 и ассоциированный Ltv1 пептид блокируют доступ rpS10; скорее всего, лишь N-терминальный домен rpS3 инкорпорируется в эти структуры. Pno1 не только соединяется с 3' концом 20S pre-rRNA, чтобы помогать расщеплению в D сайте с помощью гибко прикрепленного Nob1, но также и предупреждает от преждевременной ассоциации с rpS26, белком, который интегрируется с помощью его соотв. шаперона Tsr2 (refs157,158,162).
Очень поздние ступени сборки pre-40S включают тестирование функции субъединицы. При этом диссоциация таких факторов, как Tsr1 и Rio2, как полагают, предшествует ассоциации Rio1 с pre-40S частицей 163,164. Вместе со зрелой субъединицей 60S, фактор инициации трансляция эукариот 5B и ATPase Fap7 способствуют формированию 80S-подобных частиц, которые используются, для функциональной корректуры pre-40S частиц, активируют Nob1 для расщепления по D сайту и удаления поздних факторов сборки, таких как Nob1 и Pno1 (refs165-167) (Fig. 2).
Ribosome assembly and human diseases
В сравнении со сборкой рибосом у дрожжей, значительно меньше известно о том, как рибосомы собираются в клетках млекопитающих168. Широкий набор факторов сборки, участвующих в синтезе рибосом человека, был идентифицирован при крупно-масштабном скрининге169-173, который выявил множество гомологов с белками дрожжей, а также новые потенциальные факторы сборки рибосом.
Дефекты сборки рибосом у людей ассоциируют с раковыми опухолями174 и с рибосомопатиями, которые представлены гетерогенным набором болезней, в которых мутации рибосомных белков или факторов сборки вызывают разные фенотипические отклонения175,176. Рибосомопатии включают Diamond-Blackfan anaemia (DBA)177,178, изолированную врожденную асплению179, North American Indian child cirrhosis180, chromosome 5q- синдром181, Treacher Collins синдром182 и Shwachman-Bodian-Diamond синдром176,183, среди прочих. Несмотря на различия в их фенотипах, биологическая подноготная этих болезней может быть грубо подразделена на две группы. Первая связана с запуском p53-зависимой стрессовой реакции, а вторая являются ткане-специфическим эффектом, наблюдаемым в клонах клеток, происходящих из костного мозга или скелетных тканей184.
Активация p53 обусловлена дефектами сборки рибосом, возникающих в комплексах, содержащих рибосомные 5S RNP185-188. Важно, что реакция p53 является ключевой причиной фенотипа Treacher Collins синдрома189, chromosome 5q- синдрома190 и North American Indian child cirrhosis191.
Мутации рибосомных белков вызывают их гаплонедостаточность. В линиях клеток человека истощение рибосомных белков может приводить к накоплению определенных рибосомных предшественников192. Хотя огромное количество мутаций при DBA ассоциировано с рибосомными белками193, дополнительные мутации были найдены в rpS26 шапероне Tsr2 (ref.194).
Довольно неожиданное наблюдение связано с ткане-специфическим фенотипом DBA и др. рибосомопатий. Первоначальные гипотезы предполагали присутствие специализированных рибосом в этих тканях, которые д. транслировать специфические мРНК 195; совсем недавно была предложена модель, что ограничение количества рибосом может влиять на определенные наборы мРНК, которые необходимы для дифференцировки определенных тканей 184. Ключевым доказательством этой модели стала идентификация мутаций в специфическом для гематопоэза транскрипционном факторе GATA1 у индивидов с DBA и демонстрация, что экспрессия GATA1 может , по крайней мере, частично быть компенсирована за счет гаплонедостаточности rpS19 (refs196,197). Совсем недавние данные показали, что мутации рибосомных белков уменьшают пул доступных рибосом при DBA, это специфически воздействует на трансляцию пула транскриптов, которые необходимы для детерминации эритроидного клона 198. Мы полагаем, что это может быть общим механизмом, приложимым и к др. рибосомопатиям, таким как врожденная asplenia 179, при которой селезенка не образуется в результате гаплонедостаточности
rpSA.
Conclusions and future perspective
Eukaryotic ribosome assembly is a highly complex and tightly regulated process, the basic principles of which we are beginning to understand. Recurring themes in ribosome assembly include the systematic compaction of pre-rRNA; an enforced chronology of assembly factor binding, which is mediated by molecular mimicry and steric hindrance; and the irreversibility of key checkpoints and proofreading of functional centres. Recent advances in cryo-EM have provided detailed molecular snapshots of pre-ribosomal particles at near-atomic resolution56,57,103-108,142,157,158. The combination of cryo-EM with crosslinking and mass spectrometry57,103,199 has enabled many protein factors to be assigned. Current models of ribosome assembly are attempts to rationalize structural, biochemical and genetic data but will require regular revision in the near future as further data become available.
The dynamic and flexible nature of early ribosome assembly events together with the poorly understood roles of DEAD-box and DEAH-box helicases, GTPases, AAA-ATPases and other ATPases highlight areas in which work and the implementation of new technologies will be essential. The use of small-molecule inhibitors has already facilitated the study of key enzymatic steps in eukaryotic ribosome assembly200-202, and the identification and development of more inhibitors will be essential to 'trap' and study particular particles.
Beyond the assembly mechanism, the interplay between ribosome assembly, nutrient availability and cell proliferation is only beginning to emerge203-206. Similarly, fairly little is known about the interdependence of ribosome assembly and nucleolar morphology207. We anticipate that with more molecular snapshots of pre-ribosomal particles in combination with cell biology, super-resolution microscopy and cryo-electron tomography, the regulation and fine-tuning of ribosome assembly will come further into focus.
|
 Fig. 1: Pre-rRNA processing in yeast.
Fig. 1: Pre-rRNA processing in yeast.  Fig. 2: Assembly of the small ribosomal subunit.
Fig. 2: Assembly of the small ribosomal subunit.  Fig. 3: Cryo-electron microscopy structure of a nucleolar small-subunit precursor.
Fig. 3: Cryo-electron microscopy structure of a nucleolar small-subunit precursor.  Fig. 4: Assembly of the large ribosomal subunit.
Fig. 4: Assembly of the large ribosomal subunit.  Fig. 5: Cryo-electron microscopy structures of large-subunit precursors.
Fig. 5: Cryo-electron microscopy structures of large-subunit precursors.  Fig. 6: Structural changes occurring in nucleolar and nuclear ribosomal precursors.
Fig. 6: Structural changes occurring in nucleolar and nuclear ribosomal precursors.