Посещений: 
ТРАНС-ДИФФЕРЕНЦИРОВКА OHC в IHC
Роль гена INSM1
Trans-differentiation of outer hair cells into inner hair cells in the absence of INSM1 Teerawat Wiwatpanit, Sarah M. Lorenzen, Jorge A. Cantu,et al.
 Letter Nature (2018)| Published: 10 October 2018 DOI
https://doi.org/10.1038/s41586-018-0570-8
| |
|
Слуховая улитка млекопитающих состоит из двух типов механосенсорных волосковых клеток, отличающихся и обладающих критическими функциями для слуха. Inner hair cells (IHCs), которые представляют собой тщательно разработанный пресинаптический аппарат, передающий сигналы к нейронам улитки и передающий звуковую информацию в головной мозг. Outer hair cells (OHCs) механически умножают индуцируемые звуком вибрации, обеспечивая усиление чувствительности к звуку и точную настройку. Волосковые клетки улитки генерируются исключительно во время развития, а гибель волосковых клеток - чаще всего OHCs - является наиболее распространенной причиной глухоты. OHCs и IHCs совместно с поддерживающими клетками возникают у эмбрионов из просенсорного региона отоциста, но как волосковые клетки дифференцируются в два разных типа, неизвестно1-3. Здесь мы установили, что Insm1, кодирующий белок цинковые пальчики, который временно экспрессируется в формирующихся OHCs, укрепляет их судьбу, предотвращая транс-дифференцировку в IHCs. In В отсутствие INSM1, многие волосковые клетки, которые зарождаются как OHCs, переключают свои судьбы, чтобы стать зрелыми IHCs. Для идентификации генетических механизмов, с помощью которых оперирует Insm1 , bsd сравнивали транскриптомы незрелых IHCs и OHCs, и OHCs в присутствии и отсутствии INSM1. В OHCs, которые лишены INSM1, активируется ряд генов, большинство из которых преимущественно экспрессируются в IHCs. Гомеозисная трансформация клеток OHCs в отсутствии INSM1 в IHCs указывает на механизм, с помощью которого эти соседние механосенсорные клетки начинают отличаться: INSM1 репрессирует основной набор генов, характерный для ранних IHC, в OHCs эмбрионов и делает их нечувствительными к градиенту, индуцирующему IHC, так что они превращаются в зрелые OHCs. Без INSM1, некоторые из OHCs, в которых эти немногие характерные для IHC транскрипты активируются, транс-дифференцируются в IHCs, указывая на гены кандидаты IHC-специфической дифференцировки.
OHCs экспрессируют Insm1 временно с начала дифференцировки (эмбриональный день 15.5 (E15.5)) до приблизительно 2 дня постнатального развития(P2) 4. Нейрональные предшественники и нейроны, формирующие спиральный ганглий, также экспрессируют Insm14. Поскольку мыши, у которых Insm1 находится в полном нокауте, погибают на ст. E19.5 5,6, мы создали аллель (Insm1F), в котором вся кодирующая последовательность может быть делетирована (Fig. 1a, Extended Data Fig. 1). Мы специальными условиями устраняли Insm1F с помощью Atoh1Cre, экспрессирующегося со ст. E13.5 (на 3 дня раньшe Insm1), и происходили рекомбинации в большинстве слуховых волосковых клеток и в некоторых поддерживающих клетках, но не в нейронах спирального ганглия 7. Мы ткже устраняли Insm1F с помощью TgPax2Cre, экспрессирующегося раньше в отоцистах, и вызывали рекомбинацию в большинстве внутренних волосковых клеток 8. У мышей Insm1 устранялся до начала экспрессии в OHCs (Extended Data Fig. 2).И Atoh1Cre/+;Insm1F/F, и TgPax2Cre/+;Insm1F/F (cKO) мыши обнаруживали альтерации в порогах auditory brainstem response (ABR), это может быть связано со сдвигами в distortion product otoacoustic emissions (DPOAEs), характеристикой дисфункции OHC (Fig. 1b-d, Extended Data Fig. 3a, b). В Кортиевом органе таких мышей многие клетки в позициях OHCs (наружный компартмент) обладали признаками IHCs. Он имели крупные стереоцилии, подобные таковым у IHCs, а не более короткие, W-образные стереоцилии OHCs (Fig. 1e, f); экспрессировали характерный для IHC calcium buffer calmodulin и были лишены, специфического для OHC oncomodulin (Fig. 1e, f, h, Extended Data Fig. 3f); экспрессировали vesicular glutamate transporter 3 (VGLUT3), необходимый для пресинаптической функции IHC, и у них отсутствовал prestin, необходимый для электромобильности OHC (Fig. 1g, m); они приобретали фляго-подобную форму IHCs скорее, чем цилиндрическую форму OHCs; они имели крупные ядра как у IHCs, вместо мелких ядер OHCs (Fig. 1i, j, Extended Data Fig. 3j). Эти ядра обладали транскрипционным фактором CtBP2, который обычно экспрессируется в IHCs (Fig. 1k), и клетки содержали ряд пресинаптических лентовидных синапсов (10.6 ± 2.1 (mean ± s.d.), n = 3 mice, 39 cells) похожих на те, что обнаруживались в контрольных IHCs (16.3 ± 0.7, n = 3 littermate controls, 30 cells), вместо малого коичества таковых у OHCs (1.8 ± 0.2, n = 3 mice, 90 cells) (Fig. 1k). За редким исключением (Fig. 1g), эти аномальные клетки обладали всеми признаками IHC и были лишены таковых, характерных для OHCs, поэтому они были названы oc-IHCs (outer compartment IHCs).
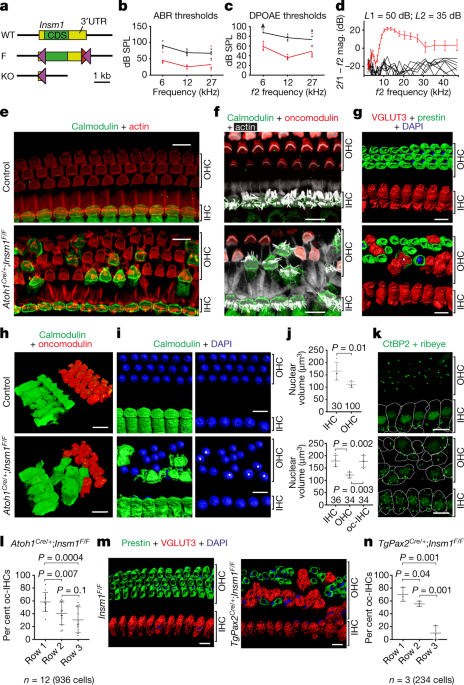 Fig. 1: Conditional ablation of Insm1 in hair cells results in IHC-like cells in place of OHCs.
Fig. 1: Conditional ablation of Insm1 in hair cells results in IHC-like cells in place of OHCs.
a, Wild-type (WT), floxed (F) and conditionally deleted (cKO) alleles of Insm1. Co-expression with Cre recombinase generates an Insm1 knockout allele lacking its coding sequence (CDS) and 3 untranslated region (UTR), leaving only part of the 5 UTR. Purple triangles, LoxP sites; red triangles, Frt sites. b-d, Hearing tests (b, c, mean with all values; d, mean ± s.e.m.). ABR thresholds (b), DPOAE thresholds (c), and iso-input functions (d) for the DPOAE of Atoh1Cre/+;Insm1F/F mice at P25-31 (black; n = 3 males and 2 females) and control littermates (red; n = 4 Insm1F/F and 4 Atoh1Cre/+;Insm1F/+; 6 males and 2 females). Up arrow (c) indicates that maximum sound was insufficient to reach threshold. e-l, Immunohistochemistry in organs of Corti revealed that the Atoh1Cre/+;Insm1F/F mice had normal IHCs but many OHCs expressed calmodulin and not oncomodulin (e, f, h), had stereocilliary bundles resembling those of IHCs (f-actin labelled with phalloidin; e, f), expressed VGLUT3 instead of prestin (g; asterisk, one rare cell expressed both), had cell shapes (h) and large nuclei like IHCs (i, j, asterisks). j, Number of nuclei measured for each cell type are indicated under each data set. k, oc-IHCs had nuclear CtBP2 and a number of presynaptic ribbons (ribeye) approaching that of IHCs. l, In Atoh1Cre/+;Insm1F/F mice, oc-IHCs are more frequent in the first row of OHCs (closer to the IHCs) than the second or third rows. Data from two males, three females and seven undetermined (936 cells). m, n, Similar distribution of oc-IHCs in TgPax2Cre/+;Insm1F/F mice. n = 3 mice (234 cells). j, l, n, Mean ± s.e.m., one-tailed Student's t-tests, n = number of mice. Images and quantifications from mid-cochlear positions at P34 (e, g, i), P46 (f, h), P21, P23 and P46 (k), and P14 and P15 (m). Controls were Insm1F/F (e, g, i, m) or Atoh1Cre;Insm1F/+ (f, j, k) littermates. Scale bars, 10 µm. Biological replicates were used for all experiments and similar results were obtained from three or more mice per genotype.
Пропорция oc-IHCs у Atoh1Cre/+;Insm1F/F мышей (42.6 ± 10.9%, n = 12 mice) и TgPax2Cre/+;Insm1F/F mice (46.0 ± 5.64%, n = 3 mice) составляла примерно половину от оставшихся видимых OHCs. Это не было связано с неполнотой или задержкой устранения Insm1, поскольку мы не обнаруживали Insm1 мРНК в оставшихся OHCs у TgPax2Cre/+;Insm1F/F мышей во время или после начала экспрессии (E16.5; Extended Data Fig. 2a, b, bottom). Очевидно, что oc-IHCs преобладали в первом ряду волосковых клеток наружного компартмента, чем во втором и третьем рядах (Fig. 1l, n). В принципе, эти oc-IHCs в зрелом органе Корти, лишенные INSM1, могут быть замещены IHCs, что вновь сгенерированные IHCs замещают потерянные OHCs, IHCs, рожденными в наружном компартменте или OHCs, которые транс-дифференцируются в IHCs. Но они не могут быть замещены IHCs, поскольку ряд IHC у обоих типов cKO мышей имеет нормальный порядок и плотность IHCs (Extended Data Figs. 3c, 4a). Хотя пво время нормального развития волосковые клетки улитки все зарождаются во время эмбриогенеза (E12-E16)9,10, конечно ранняя гибель волосковых клеток может запускать генерацию волосковых клеток благодаря пролиферации и трансдифференцировке поддерживающих клеток в первые несколько дней после родов11-15. Этого не происходит в отсутствие INSM1, т.к. плотность волосковых клеток в наружном компартменте (OHCs + oc-IHCs) не менялась до ст. P34 (Extended Data Figs. 3d, e, 4b, c), тогда как oc-IHCs уже присутствовали до этого. t Во-вторых, волосковые клетки, которые возникают постнатально из поддерживающих клеток, первоначально экспрессируют SOX212-14, тогда как ни одна из oc-IHCs у cKO детенышей не экспрессирует SOX2 (Extended Data Fig. 4d). В-третьих, некоторые постнатально продуцируемые HCs возникают в результате пролиферации поддерживающих клеток11-14, но ни одна из oc-IHCs у cKO мышей не возникала в результате постнатальной пролиферации (Extended Data Fig. 4e). Эти результаты показывают. что oc-IHCs не являются результатом гибели OHC, сопровождаемой замещением смещающихся IHCs или постнатально генерированных волосковых клеток. Вместо этого oc-IHCs представляют собой гомеотическую трансформацию (механосенсорных OHCs в IHCs) из-за генетического дефекта в генерации или дифференцировке OHCs. Любая IHCs возникшая на месте OHCs это трансдифференцировка OHCs в IHCs.
Мы исследовали орган Корти мышей после специфической обработки (TgPax2Cre/+;Insm1F/F и Atoh1Cre/+;Insm1F/F) или у полностью (Insm1GFP.Cre/-) Insm1 нокаутов во время позднего эмбриогенеза, когда начинается дифференцировка OHCs и IHCs. На ст. E16.5, все клетки наружного компартмента начинают экспрессировать самые ранние маркеры OHCs: Insm1 промотор у Insm1GFP.Cre/- эмбрионов (который лишен INSM1, но экспрессирует GFP с Insm1 промотора 4; Fig. 2a), и BCL11B в ядрах (Fig. 2b, c). В то время как у контрольных мышей экспрессия BCL11B сохраняется после рождения, у эмбрионов, лишенных INSM1, уменьшается почти наполовину в наружном компартменте волосковых клеток (oc-HCs) (с E18.5 по P2; Fig. 2b, c). Во время того же самого периода многие oc-HCs экспрессируют ранний IHC маркер fgf8 (Fig. 2d). Примерно к моменту рождения начинаю экспрессироваться два дополнительных маркера у контрольных мышей: neuroplastin, преимущественно в OHC стереоцилиях 16, и VGLUT3 в IHCs 17.При сравнении у обоих cKO мышей большинство oc-HCs экспрессируют VGLUT3 , но не neuroplastin (Fig. 2f, g). Наконец, хотя ориентация IHCs и OHCs сохраняется при рождении у cKO мышей, наблюдается дезорганизация рядов OHC, на уровне ядер уже выявляются альтерации в форме клеток (Fig. 2e). Мы полагаем, что в отсутствие INSM1 oc-HCs генерируются с ранними признаками OHC, но вскоре после этого некоторые из этих клеток теряют свои признаки, экспрессируют ранние IHC маркеры и начинают дифференцироваться в зрелые IHCs. Эта трансдифференцировка ранних OHCs в IHCs показывает, что INSM1 не нужен, чтобы инициировать выбор клеточной судьбы OHC, но действует после этого, чтобы предупредить переключение на IHCs. Insm1 действует путем консолидации судеб OHC, делая этот выбор устойчивым.
 Fig. 2: Trans-differentiation of embryonic OHCs into embryonic IHCs in the absence of INSM1.
Fig. 2: Trans-differentiation of embryonic OHCs into embryonic IHCs in the absence of INSM1.
a, GFP on embryonic organs of Corti indicates that the Insm1 promoter (expressing GFP.Cre) was active in oc-HCs (OHCs) at E16.5 and E18.5, regardless of the presence (Insm1GFP.Cre/+) or absence (Insm1GFP.Cre/-) of INSM1. b, c, Immunohistochemistry at E16.5-P2. While Bcl11b, an immature OHC-specific transcription factor, was found in all oc-HCs from control Insm1GFP.Cre/+ and Insm1GFP.Cre/- mice at E16.5 (b), its expression was diminished or undetectable (asterisks) in about half of oc-HCs in mice lacking INSM1 at E18.5 (b) and P2 (c). d, In situ hybridization revealed that in the absence of INSM1, a subset of oc-HCs began to express Fgf8 weakly as early as E16.5 (32.5%, 110/338 OHCs from n = 3 TgPax2Cre/+;Insm1F/F or Atoh1Cre/+;Insm1F/F mice) and strongly by E17.5-E18.5 (40%, 52/130 OHCs from n = 2 Insm1GFP.Cre/- or Atoh1Cre/+;Insm1F/F mice) and P0-P4 (46.6%, 61/131 OHCs from n = 3 TgPax2Cre/+;Insm1F/F or Atoh1Cre/+;Insm1F/F mice). e, Immunohistochemistry at P0 revealed that although in Atoh1Cre/+;Insm1F/F mice there was no HC loss and OHCs retained their characteristic inclination, their cell bodies were disorganized at the nuclear level. f, All OHCs at the bases of cochleae from Insm1F/F mice had neuroplastin in stereocilia (visualized with phalloidin). However, some OHCs at the base of cochleae from TgPax2Cre/+;Insm1Flox/Flox mice lacked neuroplastin. g, Conversely, the oc-HCs that lacked neuroplastin expressed VGLUT3. Images are from mid (a-e) or basal (f, g) cochleae. Scale bars, 10 µm. Biological replicates were used for all experiments and similar results obtained from three or more mice per genotype. .
Кратковременная экспрессия Insm1 достаточна, чтобы избегнуть фенотипического превращения (Extended Data Fig. 5). Очевидно, что Insm1 закрепляет судьбы OHC в течение короткого онтогенетического периода. Удивительно, что хотя KO OHCs полностью лишены Insm1 с момента своего рождения, все же меньше половины этих клеток транс-дифференцируются в IHCs. Такая транс-дифференцировка у TgPax2Cre/+;Insm1F/F и Atoh1Cre/+;Insm1F/F мышей более часта в рядах волосковых клеток, близких к IHCs, чем в тех, что расположены дальше от них (Fig. 1l, n). Такое распределение указывает на существование градиента вдоль neural to abneural оси органа Корти, которые регулирует возникновение типов волосковых клеток. Этот градиент может индуцировать дифференцировку IHC, а INSM1 может действовать, чтобы предупреждать эмбриональные OHCs от реакции на градиент.
В др. развивающихся типах клеток, INSM1 функционирует в качестве транскрипционного активатора или репрессора 18-24. Мы предполагаем, что INSM1 направляет OHCs, чтобы они развивались по другому по сравнению с IHCs путем активации OHC-специфических генов или ингибируя IHC-специфические гены. Мы впервые установили, какие гены экспрессируются в каждом типе дифференцированных волосковых клеток, когда экспрессируется Insm1 (Fig. 3), и затем исследовали гены, регулируемые с помощью INSM1 в развивающихся OHCs (Fig. 4). В обоих подходах мы использовали Insm1GFP.Cre, в котором кодирующая последовательность Insm1 замещена на ту, что дает слитый белок GFP и Cre recombinase, тем самым служит в качестве репортера, а также нулевого аллеля 4,23. Мы создали Insm1GFP.Cre/+;Atoh1A1GFP/+;R26R tdTomato/+ мышей, у которые все волосковые клетки экспрессировали GFP (начиная со ст. E13.5 с Atoh1A1GFP и в OHCs с Insm1GFP.Cre), но только OHCs экспрессировали также tdTomato вследствие экспрессии Insm1GFP.Cre (во всей улитки с E18.5 4; Fig. 3a, b). Мы использовали таких мышей, чтобы выделять OHCs и IHCs у новорожденных (P0, приблизительно E19.5) из органа Корти (Fig. 3c). Используя fluorescence-activated cell sorting (FACS), мы собирали пуля РНК из IHCs и OHCs (Fig. 3d), и затем использовали РНК секвенирование (RNA-seq) чтобы получить их транскриптомы (Supplementary Table 1). Т.о., мы идентифицировали 922 IHC-характерных генов и 676 OHC-обогащенных генов (Fig. 3e, Supplementary Tables 1-3). Среди них было 12 генов, ранее известных как экспрессирующиеся преимущественно в ранних IHCs или OHCs 4,17,25-29 (Fig. 3e), демонстрируя, что наш подход выявляет большинство дифференциально экспрессирующихся генов. Основная проблема связана с генами, которые обнаруживают незначительные отличия в экспрессии (в 2 раза или меньше; напр., Zmat3), или с теми, которые обнаруживаются на очень низких уровнях только в одном из типов клеток (напр., Sox18 и Msx1), действительно ли они дифференциально экспрессируются (Fig. 3e), а использовали quantitative PCR with reverse transcription (RT-qPCR) для тестирования дифференциальной экспрессии с использованием дополнительных пулов РНК из IHCs и OHCs. Подтверждено, что все 21 ген дифференциально экспрессируются и различия в экспрессии были сходны с теми, что были подсчитана с помощью RNA-seq или RT-qPCR (Fig. 3f). Мы также подтвердили дифференциальную экспрессию дополнительных генов с помощью метода, не чувствительного к потенциальным артефактам клеточной сортировки и экстракции мРНК: Bcl11b в OHCs с помощью иммуногистохимии (Fig. 2b); Insm2 в OHCs с использованием Insm2LacZ линии мышей (Fig. 3e inset); и др. гены с помощью гибридизации in situ (ISH) как преимущественно экспрессируемые в OHCs (Neurod6, Sez6l) или IHCs (Tbx2, Id4, Rprm, Smad3, Car13, Brip1, Lrrn1, Pink1) (Fig. 4g-o). Эти результаты свидетельствуют о низком преобладании ложно позитивных результатов среди генов, определенных нами как дифференциально экспрессирующиеся между незрелыми IHCs и OHCs.

Fig. 3: Genes preferentially expressed in immature IHCs or OHCs.
a-d, Strategy for collecting separate pools of perinatal (P0) OHCs and IHCs by FACS. a, Crosses for generating pups in which live IHC and OHCs can be fluorescently distinguished (green, GFP; red, tdTomato). b, Insm1GFP.Cre/+;Atoh1A1GFP/+;R26RtdTomato/+ mice express GFP in IHCs and GFP plus tdTomato in OHCs. Neurons express tdTomato but GFP from Insm1GFP.Cre has subsided. c, FACS separating IHCs (green) from OHCs (yellow) and neurons (red), done on six separate pools of IHCs and OHCs. d, RT-qPCR for IHC-specific Fgf8 and OHC-specific Insm1 confirm that these pools of cells are enriched for IHCs or OHCs, respectively. e, Logarithmic plot of genes preferentially expressed in either IHCs or OHCs based on RNA-seq values. Blue arrows indicate genes previously known to be HC subtype-specific in neonates4,17,25,26,27,28,29. We additionally confirmed perinatal OHC- or IHC-specific expression of Insm2 with a knock-in reporter line (E17.5; top inset). f, Comparison of fold difference in mRNA expression determined (for genes indicated as black boxes in e) by RNA-seq and RT-qPCR. Genes that encode transcription factors are underlined. IHC-specific genes potentially inhibited by INSM1 in embryonic OHCs are in blue. g, Venn diagrams indicating the number of genes enriched in either IHCs or OHCs of neonates versus adults (estimated from published results30). Representative genes are shown in parentheses. Although neonatal HCs of either type begin to show expression of some functional markers characteristic of mature cells (Otof and Slc17a8 (also known as Vglut3) in IHCs and Slc26a5 (also known as Prestin) in OHCs), the majority of hair cell type-specific genes at this early stage differ from those of the mature cells. Biological replicates were used for all experiments and similar results obtained from three or more mice per genotype.

Fig. 4: Insm1 prevents expression of a subset of immature IHC-specific genes in embryonic OHCs.
a, Identification of genes potentially regulated by INSM1 in embryonic to perinatal OHCs. Plot of average expression levels determined by RNA-seq from E18.5 OHCs expressing (Insm1GFP.Cre/+) or lacking (Insm1GFP.Cre/-) INSM1 (n = 3 pools of OHCs per genotype, each from 8-12 mice). Undetected transcripts are assigned an expression of 10 1. We established as cutoff either an FDR-adjusted P < 0.05 (red squares) or, for a less stringent selection, a raw P < 0.01 (green squares). Blue diamonds represent all other transcripts. b-e, Venn diagrams indicating overlap between IHC- or OHC-enriched genes with genes that are presumably regulated by INSM1 in OHCs. Upregulated and downregulated genes are those overexpressed in OHCs with or without INSM1, respectively. The expected number of genes that would appear by random coincidence is in parentheses. Only e shows a larger overlap than randomly expected, pointing to 36 IHC-specific genes that appear to be downregulated by INSM1 in OHCs. f, Plot of the differential expression of genes in OHCs versus IHCs and OHCs with INSM versus OHCs without INSM1. Genes are plotted as dots with colours corresponding to the P value criteria used in a. Expression levels in OHCs with and without INSM1 (from which KO/WT changes are estimated) are average RNA-seq values of three pools of OHCs per genotype. Expression levels in IHCs and OHCs (from which IHCs/OHCs changes are estimated) are average RNA-seq values of six pools of each cell type. Blue dots along the x-axis are examples of the many genes enriched in either IHCs or OHCs that are not affected by INSM1. Differentially expressed genes confirmed by RT-qPCR are labelled in purple. Each gene is upregulated to a similar extent in IHCs (versus OHCs) as in OHCs lacking INSM1 (versus IHCs with INSM1). g, Graphic interpretation. Darker shades of blue indicate higher expression. h-o, ISH confirms preferential expression in IHCs (and often other cells of the organ of Corti) compared with OHCs, and increased expression in OHCs lacking INSM1. Scale bars, 10 µm. Biological replicates were used for all experiments and similar results obtained from three or more mice per genotype.
Получены транскриптомы перинатальных волосковых клеток улитки и поддерживающих клеток, но они включают смеси и OHCs и IHCs25,26. Хотя специфичные для клеточного типа транскриптомы зрелых IHCs и OHCs, полученные с использованием microarrays, были опубликованы30, мы получили транскриптомы этих типов клеток до созревания во время ранней дифференцировки. Сравнение генов, экспрессирующихся дифференциально и в зрелых IHCs и OHCs, выявили очень немного перекрывания (Fig. 3g and Supplementary Table 4): только 5.9% из IHC-обогащенных и 2% из OHC-обогащенных генов дифференциально экспрессируются на дифференцированных и зрелых стадиях. Сюда входят некоторые гены, которые являются характерными для зрелой стадии (Vglut3 и Otof в IHCs и Prestin в OHCs) , чья экспрессия начинается при рождении. Однако, огромное большинство генов, которые предпочтительно экспрессировались в каждом типе клеток во время дифференцировки (такие как Insm1, Insm2 и Bcl11b в OHCs, и Brip1, Car13 и Fgf8 в IHCs) , не экспрессировались после созревания и наоборот. Т.о., сложные транскриптомы с участием сотен генов, временно активны во время OHC- и IHC-специфической дифференцировки. И в этом генетическом контексте INSM1 закрепляет судьбу OHCs, так что они дифференцируются в зрелые OHCs, а не в IHCs.
Чтобы исследовать, как INSM1 управляет дифференцировкой OHC, мы использовали RNA-seq, чтобы обнаружить гены, которые дифференциально экспрессируются в дифференцирующихся OHCs в присутствии и отсутствии INSM1 (Insm1GFP.Cre/+ против Insm1GFP.Cre/-) (Fig. 4a, Extended Data Fig. 6, Supplementary Table 5). Мы идентифицировали между 31 и 331 генами, которые могут дифференциально экспрессироваться (активироваться или подавляться) с помощью INSM1 (Supplementary Tables 6, 7). Сравнение этих генов с теми, которые обычно преобладают в OHCs или IHCs (Fig. 4b-e), в комбинации с повторной проверкой RT-qPCR (Supplementary Table 8) и ISH (Fig. 4f-o), показало, что в OHCs, INSM1 не активирует OHC гены, а скорее подавляет IHC гены. Не было обнаружено вновь активируемых генов с помощью RT-qPCR , а из 22 подавляемых генов 21 обычно экспрессируется преимущественно в IHCs. Преобладание этих генов these в дикого типа IHCs сходно с их активацией в OHCs , лишенных INSM1 (Fig. 4f, g, Extended Data Table 1). Напротив, большинство генов, которые дифференциально экспрессируются в OHCs в противовес IHCs не были затронуты INSM1. Мы полагаем, что INSM1 подавляет специфический набор генов, преимущественно экспрессирующихся в IHC, в эмбриональных OHCs; в отсутствие INSM1, эти гены экспрессируются в эмбриональных OHCs, половина из которых транс-дифференцируется в IHCs.
На ст. E18.5 OHCs, лишенные INSM1, имеют не активированными большинство из ранних специфичных для IHC генов и всё ещё экспрессируют ранние специфичные для OHC гены(Fig. 4f), даже если большинство из этих клеток будет в дальнейшем дифференцировано и экспрессировать все известные признаки и маркеры IHCs и не экспрессировать OHCs (Fig. 1e-k, m, Extended Data Fig. 3f). Это небольшое количество ранних специфичных для IHC генов (21 из 922, ~ 2%), которые активируются в эмбриональных OHCs, лишенных INSM1, скорее всего, представляют раннюю ступень генетического каскада, который приводит к их полной трансформации в IHCs. Т.к. oc-HCs, экспрессирующие эти немногие гены, дифференцируются в IHCs, то эти гены, скорее всего, необходимы для дифференцировки IHC. Следовательно, помимо идентификации Insm1 в качестве критического гена для дифференцировки OHC, наши результаты также идентифицировали гены канидаты на роль регуляции специфической дифференцировки IHCs. Хотя все OHCs экспрессируют Insm1, но в его отсутствие меньше половины из них транс-дифференцируются в ISH (Fig. 4g-o). Некоторые гены (Rprm, Id4, Lrrn1, Car13, Pink1 и Brip1; Fig. 4h-n) оказывались активированными во всех OHC, лишенных INSM1, следовательно, скорее всего, они обычно были репрессированы с помощью INSM1. Сюда могут входить и гены, которые не будучи подавлены с отсутствие INSM1, делают эмбриональные OHCs чувствительными к градиенту, который индуцирует транс-дифференцировку в IHC. Др. гены (Fgf8 и Tbx2; Figs. 2d, 4o) были активированы только в меньшем половины числе oc-HCs - преимущественно в тех, что будут транс-дифференцированы в IHCs. Эти гены являются одними из самых ранних экспрессирующихся в IHCs и, скорее всего, включают регуляторы дифференцировки IHC.
Наши результаты выявили гомеозистную трансформацию OHCs в IHCs в отсутствие INSM1, идентифицировали гены первоначально неправильно регулируемые в отсутствие Insm1 и установили генетический механизм дифференцировки этих двух типов клеток: зарождающиеся OHCs временно экспрессируют Insm1, который репрессирует (прямо или косвенно) стержневой набор ранних специфических для IHC генов и делает клетки нечувствительными к градиенту, индуцирующему IHC; это закрепляет судьбы OHCs путем предупреждения их транс-дифференцировку в IHCs.
|



