Посещений: 
ТРАНСЛЯЦИЯ
Структура и функция рибосом
Roadblocks and resolutions in eukaryotic translation • Anthony P. Schuller &
• Rachel Green
 Nature Reviews Molecular Cell Biologyvolume 19, pages526–541 (2018)
| |
|
Во всех доменах жизни трансляция информации, закодированной в мРНК в белок осуществлятся с помощью крупной макромолекулярной машины, наз рибосомы. Рибосомы считывают информацию одного кодона (три нуклеотида) одномоментно, транслируя в белок посредством действия tRNAs, которые распознают каждый кодон, чтобы вставить соотв. аминокислоту (Fig. 1). tRNAs действительно являются центральными 'трансляторами' в процессе трансляции.
 Fig. 1: Overview of eukaryotic translation.
Fig. 1: Overview of eukaryotic translation.
a | Translation begins with initiation, where the complex coordination of many eukaryotic translation initiation factors (eIFs), initiator methionyl-tRNA (Met-tRNAiMet), the ribosomal subunits and the mRNA to be translated come together at the AUG start codon of the open reading frame. Next, elongation involves synthesis of the peptide chain through the coordinated actions of eukaryotic elongation factors (eEFs) and aminoacyl-tRNAs (aa-tRNAs) until the ribosome reaches a termination or stop codon. In this termination phase, the peptide is released through the actions of eukaryotic peptide chain release factors (eRFs). Finally, the ribosome subunits must be recycled by ATP-binding cassette subfamily E member 1 (ABCE1) for a subsequent round of translation. b | Structural model of the yeast 80S ribosome depicting three sites for tRNA binding: E (exit; coloured orange), P (peptidyl; coloured purple) and A (aminoacyl; coloured cyan). mRNA is shown in red. The model was created by alignment of the Saccharomyces cerevisiae 80S ribosome (PDB 4V88) and Thermus thermophilus 70S ribosome (PDB 4V9I). c | Overview of the elongation cycle highlighting tRNA movement through the ribosome. tRNA selection occurs at the A site. Next, peptide-bond formation occurs, which transfers the peptide to the A-site tRNA. Concurrent with peptide-bond formation, the tRNAs adopt a 'hybrid' state (relative to the large and small ribosomal subunits), and this ribosome complex is the substrate for translocation to allow the decoding of the next codon. d | Schematic of the peptidyl-transfer reaction that occurs during translation elongation. The amino group of the incoming amino acid (cyan) attacks the ester linkage on the peptidyl-tRNA (purple) in the ribosomal P site to transfer the growing peptide chain to A-site tRNA. e | Structural model of the S. cerevisiae 80S ribosome bound to eIF5A. eIF5A binds in the ribosomal E site. Zoom-in area shows interactions between the hypusine modification of eIF5A and the CCA nucleotides at the 3? end of the peptidyl-tRNA. The model was created by alignment of the S. cerevisiae 80S ribosome (PDB 4V88) with coordinates for the 60S subunit bound by eIF5A (PDB 5GAK).
Поцесс трансляции может быть прерван в 4-х основных фазах: инициации, элонгации, завершения и рециклинга рибосом (Fig. 1a). Хотя основные аспекты трансляция очень законсервированы среди эукариот, бактерий и archaea, существуют существенные отличия в том, как каждая из этих 4-х ступеней выполняется. На первой ступени, многие эукариотические факторы инициации трансляции (eIFs) осуществляют собственно сборку 80S рибосом на AUG стартовои кодоне, при этом инициатор methionyl-tRNA соединяется с P сайтом. Во время элонгации 80S рибосомы перемещаются поступательно (processively) вдоль мРНК по три нуклеотида в каждом шаге, синтезируя кодируемый белок по одной аминокислоте в каждый момент в ходе скоординированного действия aminoacyl-tRNAs и eukaryotic elongation factors (eEFs). В ходе этого процесса tRNAs осуществляют векторный переход между тремя сайтами внутри рибосомы, который распространяется как на крупную рибосомную субъединицу (60S), так и на малую рибосомную субъединицу (40S): E сайт, P сайт и A сайт (Fig. 1b,c). В конце открытой рамки считывания (ORF), рибосома наталкивается на кодон терминации, который специфически распознается с помощью ряда белковых факторов, наз. eukaryotic peptide chain release factors (eRFs), которые способствуют высвобождению синтезируемого белка с peptidyl-tRNA (и в конечном итоге с рибосомы). Наконец, в фазе рециклинга пост-терминационный 80S рибосомный комплекс преобразуется с помощью семейства АТФ-связанных cassette subfamily E member 1 (ABCE1) на отдельные 40S и 60S субъединицы, чтобы начать новый раунд трансляции.
Каждая ступень базируется на принятии решения о дальнейшей судьбе трансляции, которое определяется состоянием рибосом, будет ли продолжаться трансляция или будет призываться механизмы контроля качества. Напр., некоторые рибосомы берут паузу, принимая решение с помощью вспомогательных факторов, таких как eIF5A, тогда как др. приостанавливаются, чтобы отказаться от трансляции с помощью рибосомы восстанавливающего фактора, такого как Dom34-elongation factor 1 α-like protein (Hbs1), комплекса (protein pelota homologue (PELO)-HBS1-like protein (HBS1L) у человека). Как и во всех процессах, кинетические события определяются с помощью концентраций в клетке соотв. факторов и предопределяют биологический исход. Такие решающие моменты могут оказывать выраженный эффект на исход от определенных мРНК путем предопределения, сколько продуктов или какие продукты будут продуцированы.
Translation elongation
Процесс элонгации начинается сражу же после инициации трансляции и как только 80S рибосома расположится на стартовом кодоне AUG с помощью methionyl-tRNA iMet на сайте P (Fig. 2a). Детали инициации трансляции описаны в нескольких обзорах 1-4. Фаза элонгации тянется с загрузки первой aminoacyl-tRNA в месте старта ORF (после инициирующего кодона) вплоть до того, когда рибосома достигнет кодона терминации в конце ORF и довольно консервативна относительно элонгации у бактерий.
 Fig. 2: Translation elongation and resolution of ribosome stalling.
Fig. 2: Translation elongation and resolution of ribosome stalling.
a | Overview of translation elongation. Aminoacyl-tRNAs are delivered to the ribosome in complex with eukaryotic elongation factor 1α (eEF1A) and GTP (not shown). Peptide-bond formation occurs, and the tRNAs are positioned in a 'hybrid' state with respect to the ribosome subunits. Subsequent translocation driven by elongation factor 2 (eEF2) causes tRNA repositioning from a hybrid state to a 'classical' state, creating an open A site for the next incoming aminoacyl-tRNA. eIF5A, which is a small protein that binds in the ribosomal E site, stimulates catalysis in the peptidyl-transferase centre throughout translation elongation. b | Ribosome stalling due to slow peptidyl-transfer kinetics (such as during the formation of Pro-Pro) is rescued by eIF5A, which promotes peptide-bond formation. c | Ribosome stalling caused by poor A-site occupancy resulting from poorly expressed tRNAs or from poor tRNA aminoacylation (not shown) can be rescued by misincorporation of near-cognate tRNAs or by frameshifting (represented as conversion of orange to purple). d | Ribosome stalling can be caused by certain consecutive tRNA-codon pair orders that are suboptimal (pink and orange) relative to synonymous pairs (purple and blue). e | Ribosome stalling caused by mRNA secondary structures can be resolved by programmed ribosomal frameshifting (PRF) at adjacent 'slippery' sequences. The example illustrates the ?1 PRF that is required for translation of the Gag-Pol fusion protein of HIV.
Элонгация при трансляции состоит из основных трех ступеней, которые проходит инкорпорация каждой аминокислоты в удлиняющуюся пептидную цепь: tRNA отбор (или декодирование), образование пептидной связи и транслокация комплекса mRNA-tRNA (Figs 1c,2a). Отбор tRNA является процессом, при котором aminoacyl-tRNA с антикодоном, соответствующим кодону мРНК, загружается на A сайт рибосомы. Aminoacyl-tRNAs доставляется на А сайт рибосомы с помощью специализированной GTPase eEF1A (elongation factor Tu (EFTu) у бактерий) в виде тройного комплекса с GTP5-7. Как только соотв. взаимодействие между кодоном и антикодоном осуществляется, то активируется eEF1A и гидролизует GTP, чтобы позволить tRNA полностью приспособиться к А сайту (rev. ref.8 для бактерий). На следующей ступени, образования пептидного мостика, амино-группа поступающей аминоислоты атакует эфирную связь на peptidyl-tRNA на P сайте рибосомы и растущая пептидная цепочка передает tRNA в A сайт (детальнее см. ref.9, у бактерий) (Fig. 1c,d). Как только сформируется пептидная связь, рибосомальные субъединицы ротируют относительно др. др. и tRNAs приспосабливается к измененной конформации, обозначаемой как 'гибридное' состояние, при котором анти-кодоновый конец tRNAs остается расположенным в значительной степени в P и A сайтах малой рибосомальной субъединицы, тогда как акцепторные концы tRNA располагаются на E сайте и P сайте крупной субъединицы (P/E и A/P состояния, соотв.)10-12 (Figs 1c,2a). Это ротированное состояние рибосомы затем становится субстратом для действия др. GTPase, eEF2 (elongation factor G (EFG) у бактерий), которая транслоцирует mRNA-tRNA комплекс относительно рибосомы и возвращает tRNAs в их 'классическое' состояние (E/E и P/P)13-17. Хотя не существует структуры для транслокации промежуточных образований (eEF2 соединяется с двумя транслоцирующимися tRNAs) у эукариот, по аналогии со структурными данными у бактерий, транслокация, как полагают, использует шарнирный поворот головки малой субъединицы и ротацию малой субъединицы относительно крупной субъединицы сопровождают перемещение tRNA в классическое E/E и P/P состояние18-20. В дополнение к eEF1A и eEF2, третий фактор, eEF3, является существенным для элонгации у грбов, потенциально способствуя высвобождению tRNA с E сайта после транслокации21,22. Такой цикл элонгации повторяется вплоть до тех пор, пока каждый кодон не будет транслирован в не будет синтезирован весь белок.
Т.к. рибосома обусловливает элонгацию вдоль ORF, она может сталкиваться с разнообразными проблематическими следствиями, которые замедляют её прогресс. Во-первых, определенные комбинации аминокислот могут останавливать рибосому или из-за плохой реакционной кинетики, обусловленной природой самих аминокислот или из-за ингибирующих конформаций синтезируемого пептида при выходе из туннеля (Fig. 2b). Последовательности мРНК, богатые кодонами для медленно экспрессирующихся определенных tRNAs также замедляют рибосомы (Fig. 2c). В некоторых случаях, порядок кодонов влияет на то, как долго рибосома сможет транслировать его, подтверждая сложность во взаимодействиях определенных tRNAs внутри рибосомы 23 (Fig. 2d). Кроме того, рибосомы могут сталкиваться с прочной вторичной структурой мРНК, такой как (стержень-петля) или псевдо-узелки, которые могут останавливать элонгацию (Fig. 2e). Во всех этих случаях рибосомы могут или продолжать или прекращать трансляцию. Некоторые из этих проблем корректируются просто за счет ожидания - рибосома ожидает до тех пор, пока соотв. фактор или tRNA не будут доставлены. Др. словами, рибосома подвергается сдвигу рамки считывания, чтобы продолжить трансляцию в др. рамке, тогда как проблематическая последовательность не является больше важной для трансляции. Если трансляция абортируется, то рибосомы преобразуются с помощью с помощью специфического аппарата. Кроме того, появляются доказательства, подтверждающие, что убиквитинирование рибосомальных белков влияет на судьбу остановки рибосом, хотя прямые механистические эффекты этих модификаций изучены плохо 24-27.
Specific amino acid combinations cause ribosome stalling
Во время элонгации рибосома обращена сталкивается с проблемой образования 400 различных пептидных связей в своем активном месте, так как любая из 20 аминокислот может быть обнаружена на Р-сайте или A-сайте tRNA. В отличие от большинства молекулярных машин, которые специфичны для одного субстрата, рибосомы могут быть достаточно поддатливыми, чтобы позволить реакцию с любым из 20 субстратов и игнорировать пертурбации в конформации peptidyl-tRNA, которые возникают в результате вновь синтезированного пептида. Хотя рибосома способна создавать все 400 из возможных пептидных связей, не реакции одинаково благоприятны (Fig. 2b). Напр., пролин является уникальным среди субстратов, поскольку его реактивный амин обнаруживается внутри кольца из 5 членов, это делает его вторичным амином со значительно пониженной нуклеофильностью. Это делает Pro-tRNA плохим акцептором peptidyl в A сайте28,29. Кроме того, благодаря энтропическим ограничениям, пролин не является идеальным донором субстратом также и в P сайте. И как результат это вносит разные негативные вклады в катализ, синтез Pro-Pro связей медленный, а добавление третьего пролина ещё сильнее затрудняет рибосомы30-33.
Хотя образования из многих пролинов кинетически медленные, участки пролинов обнаруживаются во всех геномах эукариот, а кодируемые белки, в самом деле, транслируются. Первые работы с Escherichia coli идентифицировали взаимодействующий с рибосомами белковый фактор элонгации P как важный для устранения остановки и трансляции на участках пролина 30,32,34. У эукариот гомологичный фактор eIF5A чрезвычайно распространен35 и является важным белком36, который содержит лишь 157 аминокислот и содержит уникальную пост-трансляционную модификацию, наз. hypusine37. Ранние эксперименты по фракционированию показали, что eIF5A стимулирует реакцию инициации, трансляции (образование Met-puromycin связи)38-40. В ретроспективе этот биохимический подход проще всего описать как образование пептидных мостиков, чем настоящую инициацию и многие исследования подтвердили, что преимущественной ролью eIF5A в трансляции является помощь в инициации31,41-43. В самом деле было показано, что eIF5A является критическим для рибосом эукариот для преодоления препятствий от образования Pro-Pro связи31. Эта активность зависит от уникальной hypusine модификации31, и некоторые структуры высокого разрешения предоставили первоначальные механистические детали eIF5A связывания в Е сайте рибосом44,45 (Fig. 1e). Эти структуры показали, что связывание eIF5A является несовместимым с присутствием на E-сайте tRNA, указывая тем самым, что tRNA д. быть высвобождена, прежде чем eIF5A сможет связаться. Одно из таких исследований оказалось способным отловить комплекс eIF5A-80S с присутствующим P-сайтом tRNA, тем самым установлены взаимодействия между hypusine в eIF5A и фосфатным остовом A76 нуклеотида на tRNA 3' (CCA) конце в peptidyl-transferase центре45. Эти находки подтвердили, что eIF5A способствует образованию пептидных связей путем стабилизации конформации peptidyl-tRNA для nucleophilic атаки с помощью aminoacyl-tRNA в A сайте (Fig. 1d,e).
Хотя эти исследования четко показали критическую роль eIF5A в трансляционной элонгации, участок в три или более последовательных пролиновых кодона обнаруживаются менее, чем в 10% генов дрожжей, и поэтому пока неясно, действительно ли это полностью описывает роль этого важного и чрезвычайно обильного фактора31. Недавние подходы по определению функции in vivo eIF5A с помощью беспристрастных методов, включая профилирование рибосом46 и 5PSeq47, выявили значительную приостановку для широкого спектра аминокислотных мотивов, включая те, что содержали пролин, aspartic кислоту, глицин, аланин, валин и изолейцин. Более того, эксперименты с использованием in vitro восстановленной системы трансляции, показали, что eIF5A является критическим лоя стимулирования образования пептидных связей из комбинаций аминокислот 7 из 8 проверенных комбинаций обнаружили стимуляцию с помощью eIF5A, все за исключением Phe-Phe)46. Принимая во внимание столь высокую концентрацию (>273000 молекул eIF5A на клетку у Saccharomyces cerevisiae35, что сравнимо с 630000 молекулами eEF1A35, 181000 молекулами eEF2 (ref.35) и 187000рибосомами48 на клетку) и высокое сродство к рибосомам49, eIF5A, скорее всего, вносит вклад в большую часть (если не во все) события переноса peptidyl во время трансляции. Эти данные вместе с аргументом, что eIF5A является глобальным стержневым фактором трансляции, который действует во время образования каждой пептидной связи, чтобы повысить поступательность и эффективность элонгации при трансляции (Fig. 2a).
Эти находки также подтверждают, что E сайт выполняет критическую роль в качестве сенсора рибосомальной кинетики элонгации. Вследствие образования пептидных связей и транслокации, deacylated tRNA перемещается на Е сайт рибосомы, а новая peptidyl-tRNA перемещается с A сайта на сайт P, а A сайт становится доступным для новой aminoacyl-tRNA. Предполагается, что после происходит предоставление места для следующей aminoacyl-tRNA на рибосоме, а deacylated tRNA высвобождается с E сайта, как и в случае бактериальных рибосом50. Т.о., когда образование пептидной связи замедляется (как в случае Pro-Pro), то aminoacyl-tRNA первоначально будет оставаться безучастной, но E сайт будет предсказуемо доступным для присоединения eIF5A, т.к. с E-site tRNA естественным образом диссоциирует (Fig. 1). Поэтому незанятый E сайт эффективно функционирует как сенсор для ареста трансляции и присоединение eIF5A выступает как реакция на такое состояние.
Сходная идея лежит в основе предложенной нами т.наз. аллостерической трех-сайтовой модели, которая объясняет, что оккупация E-сайта прямо влияет на точное воспроизведение трансляции в A сайте 51, хотя эта гипотеза остается дискуссионной (rev. ref.8). Сравнительно недавние наблюдения с использованием биохимии и экспериментов с Fo¨rster resonance energy transfer (FRET) одиночных молекул, в основном на бактериях, возобновили интерес к механизмам коммуникаций между оккупацией E сайта и реактивностью в А сайте 13,52,53. В частности, в недавнем исследовании было показано, что диссоциация с E сайта tRNA необходима для конформационных изменений перед транслокацией на следующий кодон 52. Итак, имеются прекрасные причины, чтобы предположить, что E сайт рибосомы является критическим модулятором кинетики трансляционной элонгации; дальнейшие исследования без сомнения приведут к усилению нашего понимания точных механизмов и эффектов подобных коммуникаций.
Codon choice affects ribosome stalling
Остановка рибосомы может также возникать, когда рибосома наталкивается на специфический редкий или субоптимальный кодон в ORF (rev.54,55). Идея редких кодонов обсуждается многие годы56, и было предположено, что организмы используют преимущественно определенные кодоны по сравнению с др., чтобы кодировать аминокислоты в определенных позициях или в определенных генах54-56. Оптимальность определенного кодона является отражением его использования в транскриптоме и доступности соотв. tRNA для использования транслирующими рибосомами57-61. Оптимальные кодоны обладают пулом tRNAs, уже доступными для элонгации; напротив, субоптимальные кодоны имеют ограниченный доступ к соотв. tRNA для трансляции56,62. В последнее время установлены корреляции между средней оптимальностью кодонов для всей мРНК и долей её трансляции 62-65. Родственная, но отличная, проблема может быть результатом неэффективного aminoacylation с помощью соотв. aminoacyl-tRNA синтетазы и следовательно снижения уровня подходящей aminoacyl-tRNA66.
Молекулярная сигнатура остановки в результате снижения уровней или tRNA или аминоацилирования является кинетическим барьером для трансляционной элонгации, возникающей в результате незанятости A сайта (Fig. 2c). Рибосома может стоять вплоть до того. как не будет доставлена соотв. tRNA на сайт A, до тех пор, пока не будет доставлена почти подходящая (near-cognate) tRNA (miscoding) или пока не случится неканоническое событие, такое как сдвиг рамки считывания67-70 (Fig. 2c). Если ни одно из этих событий не происходит, то остановившаяся рибосома может быть подвергнута контролю качества71. Др. исследование идентифицировало корреляции между общей оптимальностью мРНК и её стабильностью, которые, скорее всего, связаны с более жесткими механизмами контроля качества, которые запускаются на проблемных мРНК (подробнее см.72).
Недавнее исследование идентифицировало неожиданное легкое затруднение в кодировании мРНК, когда определенный порядок кодонов может стать проблемным, даже если ни один из кодонов не является редким. При скрининге библиотеки рандомизированных GFP, содержащей случайные кодоны в соседних позициях, идентифицированы 17 разных пар кодонов, ингибирующих трансляцию 23. В некоторых случаях ингибирование может быть объяснено низкой концентрацией определенных tRNA или, скорее всего, за счет неэффективного колеблющегося декодирования. Однако, в др. случаях авт. наблюдали, что определенный порядок кодонов в паре влияет на оптимальность трансляции (Fig. 2d). Напр., пара кодонов CUC-CCG (кодирующая Leu-Pro) сильно подавляет трансляцию по сравнению с оптимально Leu-Pro-кодирующей UUG-CCA; важно, однако, что два одних и тех же кодона в обратном порядке CCG-CUC (Pro-Leu) не являются более подавляющими. Эти наблюдения подтверждаются тонкой координацией между P-site и A-site tRNAs во время элонгации и подчеркивают потенциальную сложность генетического кода.
Ribosome stalling caused by mRNA structures
Остановка рибосомы может также возникнуть в результате образования прочной вторичной структуры в мРНК. Присутствие в РНК образования ствол-петля или псевдо-узелков, как полагают, вызывает остановку. В некоторых из этих случаев рибосомы останавливаются на верхушке повторяющейся, 'slippery' последовательности, такой как AAAAAAG, которая , в свою очередь, способствует сдвигу рамки считывания в рибосоме73-78 (Fig. 2e). Многие из таких сайтов являются продуктом естественного отбора и обозначаются как сайт запрограммированного рибосомального сдвига рамки (PRF)79. Хотя приостановка еа сайте PRFсходна с паузой, описанной выше, здесь имеется определенное преимущество в отношении ожидаемого исхода экспрессии гена.
Вирусы обычно используют PRF сайты для более эффективного кодирования генов в своем ограниченном геноме. Часто события сдвига рамки считывания контролируют соотношение транслируемых вирусных структурных и энзиматических белков, таких как в HIV-1, где -1 PRF приспосабливает соотношение Gag к Gag-Pol экспрессии, необходимое для репликации и сборки собственных вирусных частиц 80,81. Сигнал HIV-1 PRF состоит из двух частей: структуры мРНК в виде ствол-петля и в виде U-богатой скользкой (slippery) последовательности 74. Структура приводит к остановке рибосом непосредственно поверх slippery последовательности, где медленная реакционная кинетика увеличивает вероятность сдвига рамки считывания, это делает возможной трансляцию нижестоящего не вписывающегося в рамку (out-of-frame) pol гена (Fig. 2e). Снова рибосома обращена лицом к барьеру, который влияет на кинетику элонгации (в этом случае stem-loop), нов этом случае запрограммированный сдвиг рамки считывания позволяет рибосоме преодолевать барьер и продолжать трансляцию нижестоящего гена.
Translation termination
Процесс завершения трансляции начинается, когда рибосома наталкивается на стоп кодон в А сайте рибосомы 82 (Fig. 3a,b). У эукариот все три мтоп кодона (UAA, UAG и UGA) распознаются с помощью одиночного release factor, eRF1. Общая форма и размер eRF1 очень сходны с таковыми для tRNA, как и ожидалось для молекулы, которая будет соединяться с A сайтом, он обладает двумя самостоятельными функциями. Во-первых, eRF1 содержит структурный мотив Asn-Ile-Lys-Ser (NIKS) и несколько иных законсервированных элементов, включая Gly-Thr-Ser (GTS) и YxCxxxF мотивы, которые распознают три терминационных кодона, с высокой специфичностью 83-87. Во-вторых, eRF1 имеет точно позиционированный мотив Gly-Gly-Gln (GGQ), который распространяется на центр peptidyl-transferase, чтобы способствовать высвобождению синтезированного пептида 88, как и у бактериальной пептидной цепочки высвобождающих факторов RF1 и RF2 (ref.89) (Fig. 3b,c). Наконец, подобно tRNA, eRF1 нуждается в специализированной GTPase, eRF3 (ref.90), для собственной функции; eRF3 наиболее близка к eEF1A и EFTu 91. eRF3, как известно, стимулирует завершение трансляции GTPase-зависимым способом 92-94 и может быть необходим для диссоциации eRF1 после высвобождения пептида; как таковой, он является критическим, когда eRF1 присутствует на substoichiometric уровнях 95. После привлечения eRF1-eRF3 комплекса на рибосому, eRF3 диссоциирует вследствие гидролиза GTP (подобно EFTu), и мотив GGQ в eRF1 оказывается способным координировать молекулу воды в центре peptidyl-transferase, которая гидролизует только что синтезированный пептид от peptidyl-tRNA 88(Fig. 3c). После высвобождения пептида, процесс терминации заканчивается.
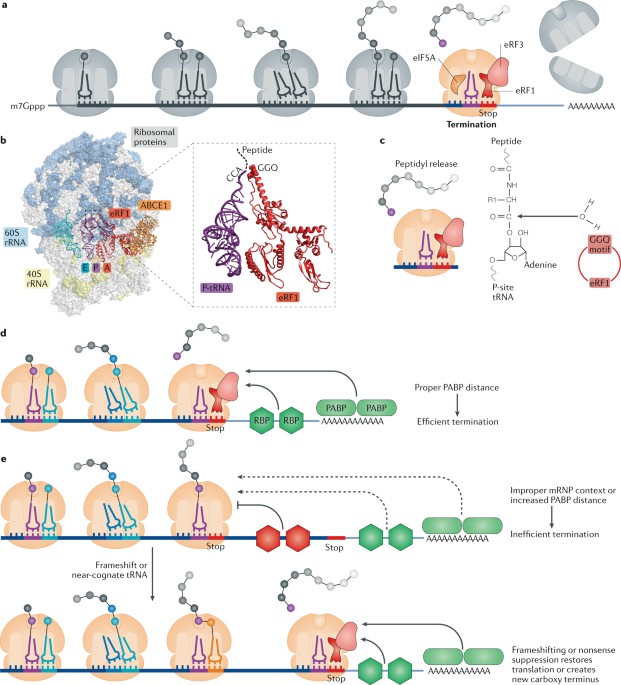 Fig. 3: Translation termination and the role of mRNP context.
Fig. 3: Translation termination and the role of mRNP context.
a | Overview of translation termination. When the ribosome encounters a termination (stop) codon, eukaryotic peptide chain release factor 1 (eRF1) is delivered by eRF3 to catalyse peptidyl hydrolysis at the ribosome peptidyl-transferase centre. eIF5A binds in the ribosomal E site to stimulate eRF1-mediated hydrolysis. b | Structure of the 80S-eRF1-ATP-binding cassette subfamily E member 1 (ABCE1) complex (PDB 3JAH) with close-up view showing the GGQ motif of eRF1, which is positioned to coordinate a water molecule for peptidyl hydrolysis of the P-site peptidyl-tRNA. c | Schematic of peptidyl-release reaction coordinated by eRF1. d | Proximity to poly(A)-binding protein (PABP) and other stimulatory RNA-binding proteins (RBPs) can affect translation termination efficiency. e | Alternatively, if a termination codon is located in a nonideal messenger ribonucleoprotein (mRNP) context, far from PABP or stimulatory RBPs, or if certain inhibitory RBPs (coloured red) are present near the stop codon, the ribosome may terminate inefficiently. In these cases, the ribosome may undergo frameshifting or incorporate a near-cognate tRNA (orange) to continue translation until a more ideal stop codon is reached.
Хотя eRF1является главным каталитическим фактором завершения трансляции, др. транс-действующ0ие факторы, по-видимому, также влияют на завершение трансляции. ABCE1 (Rli1 у S. cerevisiae) взаимодействует с eRF1 (refs. 96-98), чтобы стимулировать каталитическую активность ( kcat) eRF1 (ref.98), а cryo-EM структура ABCE1-eRF1-80S комплекса показывает, что ABCE1 стабилизирует активную конформацию eRF199 (Fig. 3b). ABCE1 является ключевым каталитическим фактором для рециклинга рибосом. Напротив, некоторые компоненты мульти-мерного фактора инициации eIF3 и ассоциированного с ним фактора eIF3J (eukaryotic translation initiation factor 3 subunit J (Hcr1) у S. cerevisiae) способствует считыванию стоп кодонов100,101, которые обычно считаются индикаторами снижения эффективности терминации.
Контекст последовательностей также влияет на эффективность завершения трансляции. Недавние структурные исследования 80S-eRF1 комплексов выявили взаимодействия между 18S rRNA и +4 нуклеотидом терминационной последовательности (первый нуклеотид 3' untranslated region (UTR))85,86. Эти структурные наблюдения согласуются с наблюдением за профилями рибосом, в которых размер ribosome protected fragments (RPFs) на стоп кодонах на рлдин нуклеотид длиннее, чем RPFs на смысловых кодонах102. Более того, роль +4 нуклеотида была подтверждена с использованием биоинформатики103 , а совсем недавно экспериментально подтверждена на дрожжах101,104,105 и клетках млекопитающих106. Эти исследования выявили, что определенные стоп кодоны и качественные особенности +4 нуклеотида более или менее склонны способствовать считыванию стоп кодона. Более слабые кодоны терминации, такие как UGAC, приводят к более высоким уровням считывания по сравнению с более сильными кодонами терминации, такими как UAAG, что было измерено по считыванию инкорпорации near-cognate aminoacyl-tRNAs107 или с помощью сдвига рамки считывания (rev. ref.108). Т.о., хотя все три стоп кодона осуществляют терминацию, +4 нуклеотид добавляет дополнительную сложность в регуляции терминации, что может влиять на общую экспрессию генов или создавать уникальные carboxy-terminal белковые расширения109,110. Появляются доказательства, подтверждающие, что помимо +4 нуклеотида, контекст последовательностей, окружающих кодон терминации (включая 3' UTR)110-114 и элементы только что синтезированного пептида46,115 также могут влиять на эффективность терминации.
Завершение трансляции механически совершенно сходно с элонгацией трансляции: в случае переноса peptidyl при элонгации, нуклеофильная атака на peptidyl-tRNA происходит с аминокислоты, конъюгированной на прибывающей на A сайт tRNA, тогда как при терминации нуклеофильная атака происходит от молекулы воды, скоординированной с помощью GGQ мотива в eRF1 (Fig. 3c). Кинетически, скорость высвобождения peptidyl медленнее, чем скорость переноса peptidyl 116, в принципе из-за того, что молекула воды является менее нуклеофильной, чем большинство аминокислот. Эти in vitro кинетические наблюдения согласуются с наблюдением in vivo, что накопление рибосом на стоп кодонах в экспериментах профилирования рибосом обычно более значительны, чем в среднем на смысловых кодонах (см. ref.46 , напр.). В недавнем исследовании, eIF5A не только сильно увеличивал скорость переноса peptidyl, но и также увеличивал скорость высвобождения peptidyl 46. Это не особенно удивительно, принимая во внимание, что химия этих двух событий весьма сходна (Figs 1d,3c). Важен, однако, тот факт, что eIF5A способствует завершению трансляции, это дополнительно подтверждает роль E сайта как сенсора медленной кинетики и как сайта, посредством которого скорость трансляции может модулироваться. В этом случае, терминация является кинетически медленной, E сайт, скорее всего, оказывается незанятым и eIF5A должен будет связаться с и стимулировать высвобождение peptidyl.
RNA-binding proteins regulate translation termination
Недавняя работа пролила свет на более тонкие механизмы, которые регулируют процесс терминации. Хотя +4 нуклеотид участвует в непосредственном взаимодействии мРНК с завершающей рибосомой, контекст стоп кодона также должен влиять на процесс терминации путем рекрутирования РНК-связывающих белков, которые взаимодействуют с release факторами или с рибосомами. Недавняя работа по poly(A)-binding protein (PABP; известен также как PABPC1) подтвердила, что это может влиять на эффективность терминации. Во-первых, физические взаимодействия между PABP и amino концом eRF3 , как было установлено, с использованием ко-иммунопреципитации и pull-down подхода117-119, имеет место, хотя последствия такго взаимодействия неясны. Использование in vitro восстановленной системы терминации показало, что PABP может прямо способствовать рекрутированию eRF1-eRF3 на terminating рибосому120 (Fig. 3d). Последующие биохимические эксперименты также выявили, что PABP стимулирует peptidyl-hydrolysis активность eRF1-eRF3, хотя механизм этого всё ещё неизвестен120.
Дополнительные связи между PABP и завершением трансляции выявлены в исследованиях пути nonsense-mediated decay (NMD . NMD является путем контроля качества, который избирательно деградирует мРНК с кодонами преждевременного окончания (PTCs). Этот путь, скорее всего, является критическим, т.к. минимизирует продукцию укороченных и часто вредных белков. Исследование, в котором PABP искусственно прикреплялся к мРНК в разных позициях, показало, что близость PABP к PTC снижает эффективность NMD, возможно путем усиления эффективности терминации или рециклинга рибосом121. Хотя в точности связи между терминацией и NMD остаются неуловимыми, эти находки подтверждают, что близость PABP или др. белков, связывающих мРНК, может влиять на скорость терминации, а значит и на распад мРНК (Box 1).
Идея, что контекст мРНК может влиять на окончание трансляции, недавно подтверждена открытием, что некоторые реснитчатые и трипаносомы могут использовать три канонических стоп кодона, чтобы кодировать аминокислоты (sense) на некоторых сайтах122-124. У некоторых из этих организмов секвенирование РНК легко идентифицирует новые tRNAs с антикодонами, которые прямо спариваются со стоп кодонами (исторически известны как супрессоры tRNAs125). Дальнейшая работа, которая включала профилирование рибосом привела к модели, согласно которой стоп кодоны распознаются с помощью aminoacyl-tRNA в качестве субстратов по умолчанию, если бы они были только размещены достаточно близко к хвосту poly(A), в этом случае они распознаются каноническими eRFs122,123 (Fig. 3d,e); стимулирующая терминацию роль PABP прекрасно вписывается в эту модель. Др. реснитчатые, по-видимому, содержат большое число стоп кодонов в рамке считывания, которым предшествует slippery последовательность, которая способствует сдвигу рамки считывания126. Снова в этих случаях исходы сдвига рамки считывания вытесняют относительно неэффективную терминацию, когда стоп кодон находится на относительно более высоком положении, терминация доминирует, если стоп кодон достаточно близок к хвосту poly(A). Подтверждение этой модели близости к poly(A) получено в наблюдении, что мРНК у этих организмов обычно содержат короткие (или несуществующие) 3' UTRs122,123. Трудности работы с этими необычными организмами не позволяют обнаружить прямую связь с PABP in vivo.
В дополнение к PABP, др. белки, связывающие мРНК, могут выполнять роль в терминации, особенно, если учитывать огромное количество белков, связывающих мРНК 127, и что многие не имеют определенной функции. Одним из таких примеров является гетерогенный ядерный рибонуклеопротеин A2/B1, который способствует считыванию мРНК для vascular endothelial growth factor A, чтобы создавать изоформу с уникальным carboxy окончанием 128. Кроме того, некоторые белки, связывающие мРНК, участвующие в пути NMD, включая nuclear polyadenylated RNA-binding protein 4 (Nab4; известен также как Hrp1) (ref.129) и poly uridylate-binding protein (Pub1) 130 у почкующихся дрожжей и apolipoprotein B mRNA-editing enzyme 1 (APOBEC1)фактор комплементации 131 и polypyrimidine tract-binding protein 1 (ref.132) в клетках млекопитающих, по-видимому, являются потенциально непосредственными регуляторами аппарата трансляции и тем самым могут положительно или отрицательно регулировать завершение трансляции.
|
Box 1 Connections between nonsense-mediated decay and mRNP context in termination and recycling
In eukaryotes, the nonsense-mediated decay (NMD) pathway selectively degrades mRNAs that contain what is broadly referred to as a premature termination codon (PTC) dependent on the three conserved regulators of nonsense transcripts UPF proteins: UPF1, UPF2 and UPF3 (refs172,173). Such mRNAs are created through multiple routes: genes may carry a mutation that results in a PTC174, inefficient splicing may lead to export of a pre-mRNA with a PTC that is (almost inevitably) encoded in the intron175,176, or the stop codons of upstream open reading frames177,178 and non-coding RNAs179,180 can be sensed as PTCs.
In many eukaryotes, the presence of a termination codon upstream of an exon junction complex (EJC; a large protein complex deposited at splice junctions) is known to be a strong signal for NMD181. UPF2 interacts with components of the EJC and with UPF1 (refs181,182), potentially recruiting UPF1 to a nearby terminating ribosome to signal NMD. However, some organisms that lack an EJC (such as Saccharomyces cerevisiae) have robust NMD, suggesting that the NMD machinery is recruited to PTCs by mechanisms independent of the EJC.
A more general mechanism of NMD may be mediated in part by the proximity of poly(A)-binding protein (PABP) to the stop codon. In budding yeast, for example, an experiment tethering PABP to an mRNA at various sites found that the proximity of PABP to a PTC affected the efficiency of NMD, likely by impacting the efficiency of translation termination or ribosome recycling121. As PABP stimulates translation termination directly120, it is possible that the distance between the PTC and PABP can determine whether the mRNA undergoes NMD or not. Indeed, PTCs located near the 3? end of open reading frames are weaker substrates for NMD183.
A broad and simple view might be that the efficiency of termination and/or recycling is crucial for distinguishing between authentic and premature stop codons, and the context of a stop codon is crucial in defining the efficiency of the termination or recycling process. This context may include proximal (or more distal) mRNA sequences or the constellation of RNA-binding proteins (RBPs) (both stimulatory and inhibitory) that are recruited to them.

|
Ribosome recycling
В конце завершения трансляции комплекс 80S, содержащий deacylated tRNA в P сайте д. быть подвергнут рециклингу в свои 40S и 60S субъединицы (Fig. 4a). Рециклинг в субъединицы осуществляется с помощью ABCE1, важного белка у всех эукариот, содержащих два домена, связывающих нуклеотиды, и amino-terminal iron-sulfur (Fe-S) кластер 133. Используя генерацию сил за счет связывания и гидролиза АТФ, ABCE1 диссоциирует рибосому после терминации на 40S и 60S субъединицы в сотрудничестве с eRF1 (refs97,98,134). Находки у S. cerevisiae также подтверждают, что ABCE1-eRF1 взаимодействуя связаны с терминацией и рециклингом, поскольку ABCE1 стимулирует гидролиз peptidyl с помощью eRF1 (ref.98). Разделенные рибосомальные субъединицы затем связываются доступными факторами инициации для следующего раунда инициации трансляции 135. После диссоциации субъединиц мРНК и тРНК также должны быть удалены с 40S субъединицы, обычно благодаря активности ligatin (известен также как eIF2D) или родственного белкового комплекса malignant T-cell amplified sequence 1 (MCTS1)-density regulated protein (DENR) 136,137.
 Fig. 4: Ribosome recycling and rescue.
Fig. 4: Ribosome recycling and rescue.
a | Overview of ribosome recycling by ATP-binding cassette subfamily E member 1 (ABCE1). ABCE1 binds to 80S ribosomes loaded with eukaryotic peptide chain release factor subunit 1 (eRF1) and uses the power generated from ATP binding and hydrolysis to dissociate the ribosomal subunits. ABCE1 remains bound to the 40S subunit to stimulate subsequent translation initiation steps. b | Superposition of ABCE1 structures in the pre-splitting (PDB 3JAH) and post-splitting (PDB 5LL6) states to highlight the 150-degree rotation of the iron-sulfur (Fe-S) cluster that occurs during ribosomal subunit dissociation. c | Superposition of ABCE1 structures pre-splitting and post-splitting, with eRF1 and ribosomal protein uL14. In the pre-splitting state, the Fe-S cluster of ABCE1 interacts with eRF1. Following splitting, the Fe-S cluster undergoes a dramatic conformational change, which drives eRF1 into the intersubunit space of the ribosome to promote subunit dissociation. In the post-splitting state, the Fe-S cluster would clash with 60S ribosomal protein uL14, thereby preventing 60S rejoining and effectively completing the recycling reaction. d | Dom34-elongation factor 1 ?-like protein (Hbs1) in coordination with ABCE1 rescues stalled ribosomes at the truncated 3? ends of mRNAs resulting from endonucleolytic or exonucleolytic cleavage or those found translating the poly(A) tail. e | Inactive or 'hibernating' Stm1-bound 80S ribosomes in Saccharomyces cerevisiae can be rescued by Dom34-Hbs1 in coordination with ABCE1 to re-enter the cytoplasmic pool of translating ribosomes.
Эксперименты по профилированию рибосом у истощенных по ABCE1 дрожжей подтвердили его роль в рециклинге рибосом in vivo и подчеркнули последствия для клетки в случает отсутствия рециклинга138. После истощения ABCE1 увеличивалась занятость рибосомами как стоп кодонов, так и 3' UTR, указывая на дефекты в терминации и/или рециклинге. Дополнительные эксперименты с использованием голодания по аминокислотам и с репортерными конструкциями показали, что , по крайней мере, в некоторых случаях рибосомы в 3' UTR активно транслировались, обнаруживая повторную инициацию трансляции вблизи стоп кодна основной ORF.
Ранее было показано, что участие в рециклинге ABCE1 первоначально было ассоциировано с инициацией трансляции. Работы нескольких лаб. показали, что ABCE1 совместно иммунопреципитируется с некоторыми факторами инициации139-141. Было установлено, что осадок с 40S субъединицами в градиенте сахарозы до некоторой степени зависит от активности ATPase139,141, а генетическое истощение ABCE1 у дрожжей нарушает сборку преиниационного комплекса, как было установлено, с помощью анализа полисом141. Т.к. процесс рециклинга рибосом происходит немедленно перед инициацией цикла трансляции, то, скорее всего, ранние и поздние исследования согласовались относительно ABCE1, относительно его важной роли в соединении последовательных процессов рециклинга и инициации.
Cryo-EM исследования структуры ABCE1, связанной с 80S рибосомами85,99 и с 40S субъединицами142 позволили предположить, как ABCE1 может функционировать, чтобы стимулировать рециклинг рибосом и рекрутирование аппарата для инициации следующего раунда трансляции. В 80S-ABCE1 структуре перед расщеплением ABCE1 Fe-S кластер располагается на А сайте рибосомы, осуществляя непосредственный контакт с С-терминальным доменом eRF185,99, тогда как в 40S-ABCE1 структуре после расщепления клвчтера Fe-S из ABCE1 принимает полностью отличающуюся конформацию, ротирует примерно на 150 градусов относительно исходного до расщепления состояния142 (Fig. 4b). Совмещение этих структур указывает, что во время расщепления перемещение домена Fe-S (координируется с помощью присоединения и/или гидролиза АТФ) сдвигает eRF1 глубже в меж-субъединичное пространство рибосом, приводя к диссоциации субъединиц. После разделения Fe-S кластер д. столкнуться с белком более крупной субъединицы рибосом uL14 (RPL23) из 60S субъединицы и тем самым предохранить 60S от воссоединения и обеспечить необратимость рециклинга (Fig. 4c).
ABCE1 был также благодаря случаю визуализирован в исследовании, описывающем структуру 48S преиниационного комплекса из клеток млекопитающих 143-145. Хотя плотность ABCE1 в этой структуре была первоначально приписана eIF3i и eIF3g, эта интерпретация позднее скорректирована с ABCE1. Важно, что эта структура включает также др. членов преиниационного комплекса, включая субъединицы eIF2 и eIF3, тем самым привнося определенный смысл тому, как ABCE1 может выполнять двойную роль в поддержании рециклинга рибосом и инициации трансляции посредством инициальных взаимодействий с пост-терминационным комплексом 80S рибосом и позднее посредством взаимодействий с 40S субъединицей и факторами инициации.
Ribosome rescue
Так как рибосомы регулярно останавливаются в ответ на препятствия, мы подозреваем, что во многих случаях остановки разрешаются продуктивно. Мы также знаем, что иногда такие механизмы неспособны и в этих случаях восстановление рибосом выступает как крайний случай. У эукариот существует сложный набор аппаратов, которые целенаправленно воздействуют на эти комплексы рибосом, чтобы деградировать синтезируемые пептиды и проблематичные мРНК и чтобы восстановить пойманные в ловушку рибосомы146,147.
Ранние генетические исследования на почкующихся дрожжах идентифицировали Dom34 (pelota у др. эукариот) в качестве критического фактора для избирательной деградации мРНК, содержащих структуры ствол-петля, и придумали феномен no-go decay (NGD)148. NGD считается как путь контроля качества мРНК, который деградирует мРНК с последовательностями, которые подавляют трансляцию, включая вторичные структуры мРНК, участки неоптимальных кодонов или укороченные мРНК148. Dom34 структурно сходен с eRF1 (refs149,150) помимо трех важных исключений: он не содержит домена, содержащего NIKS мотив, который распознает стоп кодоны; он не содержит мотив GGQ, которые катализирует peptidyl гидролиз; и он поставляется в рибосомы с помощью самостоятельной GTPase, наз. Hbs1 (refs151,152). Мы отметили, что существуют дополнительные изоформы153,154 и гомологи155 для Hbs1 у высших эукариот, которые усиливают разнообразие этой системы.
Структурное сходство с eRF1 указывает на непосредственную роль Dom34 для рибосома (скорее, чем участие в нижестоящем распаде мРНК 148), но, скорее всего, некая самостоятельная роль придает ключевые отличия, представленные выше. В самом деле, биохимические работы с использованием in vitro восстановленной системы трансляции показали, что Dom34 и ассоциированная GTPase Hbs1 диссоциируют субъединицы рибосом независимо от peptidyl высвобождения и независимо от качественных особенностей кодонов, расположенных в A сайте156 (Fig. 4d). Кроме того, Dom34-Hbs1, по-видимому, преимущественно диссоциирует комплексы рибосом, несущие виды мРНК, укороченные на своих 3' концах98,157. Важно, в то время как Dom34 сам по себе достаточен, чтобы способствовать разделению субъединиц in vitro156,, фактор рециклинга ABCE1, по-видимому, существенно увеличивает скорость диссоциации субъединиц с помощью eRF1 и Dom34 (refs97,98,157).
Хотя эти биохимические исследования подтверждают роль Dom34 в восстановлении рибосом, его мишени in vivo неизвестны. Последующие эксперименты с использованием профилирования рибосом и репортерных конструкций у дрожжей выявили, что их естественные мишени включают несколько самостоятельных видов рибосом, которые накапливаются иногда в неожиданных местах. Во-первых, Dom34 восстанавливает рибосомы, не подвергшиеся рециклингу, остановившиеся в 3' UTR,138,158 или те, которые транслируются в poly(A) хвост (non-stop decay) и здесь останавливаются159,160 (Fig. 4d). Кроме того, Dom34, по-видимому, восстанавливает остановившиеся рибосомы, обнаруживаемые на конце укороченных видов мРНК, возникших в результате эндонуклеотического (или экзонуклеотического) расщепления, которое может возникать в результате разнообразных событий остановки, включая собственно редкие кодоны и структуры ствол-петля148,160, NSD159,160 или как часть реакции на неупакованные белки158,161.
В целом эти данные подтверждают генеральную роль Dom34 в удалении рибосом, которые транслируют в проблематичные регионы. Важно, что некоторые внутренние последовательности, останавливающие рибосомы, могут не зависеть от обеспечиваемого с помощью Dom34 восстановления, включая те, которые вызываются гистидиновым голоданием158, остановкой, вызванной CGA-кодоном152 или остановкой на poly-Arg последовательности162. Впоследствии будут определены полностью специфичности Dom34 восстановления in vivo.
Принимая во внимание широту функции восстановления рибосом, Dom34 является критическим в в поддержании гомеостаза рибосом . Отсутствие Dom34 впервые было установлено в геноме дрожжей, обнаруживающих дефекты роста, возникающие в результате гаплонедостаточности рибосомального белка163. Недавние эксперименты показали, что Dom34-Hbs1 совместно с ABCE1 инактивирует рециклинг, 'вызывает бездействие' 80S рибосом, связанных супрессорным белком Stm1 и тем самым поддерживая пул компетентных к транслокации рибосом 164 (Fig. 4e). У S. cerevisiae, Stm1, ассоциируемый с не транслирующими 80S рибосомами вследствие лишения питательных веществ и играющий ключевую роль в восстановлении трансляции после стресса, удален165-167. Структурный анализ Stm1-связанных 80S рибосом показал, что Stm1 связан с каналом вступления мРНК, где он взаимодействует с 40S и 60S субъединицами, чтобы запереть это трансляционно подавленное состояние168; активность Dom34-Hbs1 необходима для диссоциации субъединиц, позволяя тем самым вернуться им к трансляции.
Эти находки согласуются с недавними наблюдениями определенных клонов клеток крови 169,170.В частности, недавнее исследование на эритроидных клетках человека свидетельствует, что необычная регуляция Dom34 (PELO у человека) экспрессии компенсирует клеточные дефекты в рециклинге рибосом, возникающие в результате потери ABCE1 (ref.170) (Box 2). Это важно для выявления динамической регуляции факторов восстановления и рециклинга, чтобы регулировать доступность рибосом в этой биологической системе. Значение этих результатов для класса болезней, известных как ribosomopathies 171 будет интересно исследовать в дальнейшем. Считается, что восстановление и рециклинг рибосом очень важны для поддержания гомеостаза рибосом во всех типах клеток
155,163,170.
|
Box 2 Implications of ribosome recycling defects for human disease
A recent study using a human erythroleukaemia cell line discovered a dynamic regulation of the ribosome rescue factors ATP-binding cassette subfamily E member 1 (ABCE1) and protein pelota homologue (PELO)-HSB1-like protein (HBS1L) that resulted in reprogramming of the cellular translation machinery170. During the haemin-mediated differentiation of K562 cells into erythrocytes, the levels of ABCE1 gradually decline, concomitant with an increase in unrecycled ribosomes at mRNA 3' untranslated regions (UTRs) (see the figure). During the initial phase of ABCE1 loss, sharp increases in the levels of the ribosome rescue factor PELO were observed at the exact point of the induction of haemoglobin expression. Following this point, PELO levels decreased, and ribosomes began to substantially accumulate at the 3' UTRs of all the mRNAs in the cell. At this stage, without ABCE1 or PELO to recycle or rescue ribosomes, the cells experienced a global decrease in ribosome availability, which ultimately led to a global translation defect and a trend towards decreased haemoglobin output.
These findings improve our understanding of ribosomopathies, which comprise a heterogeneous set of diseases that result from perturbations in ribosome homeostasis, such as the loss of a ribosomal protein, and in humans most typically present as haematopoietic dysfunction184. Diamond-Blackfan anaemia is a well-documented ribosomopathy that results from the heterozygous loss of any of a number of ribosomal proteins and is associated with a reduced erythroid progenitor cell population185. The same study that identified a dynamic regulation of ribosome rescue factors in the differentiating K562 cells also explored the connection between ribosome rescue factors and the loss of 40S ribosomal protein eS19 (RPS19)170. Importantly, overexpression of PELO- HBS1L was sufficient to rescue defects in haemoglobin synthesis that resulted from eS19 depletion. Together, these results are consistent with the idea that careful regulation of ribosome rescue factors is important for maintaining a cytoplasmic pool of active ribosomes that is suitable for supporting translation homeostasis.
![]()
|
Conclusions and future perspective
Although the ribosome was discovered more than 60 years ago and the genetic code deciphered in the 1960s, a host of new data underscore the complexity of translation and the possible layers of regulation of gene expression that exist beyond the simple three-letter codon table. As the ribosome proceeds through the phases of translation (Fig. 1), it encounters many roadblocks that can have a profound impact on the translational output of a particular mRNA. Although these problematic encounters are diverse in nature, in each case, the subsequent steps of translation are affected, and so the ribosome must either continue or abort translation. The different outcomes can have a dramatic effect on global gene expression and thus on the physiology of the organism.
During translation elongation (Fig. 2), the task of the ribosome is to synthesize the mRNA-encoded polypeptide, even when particular sequences of amino acids, codons or interactions between tRNAs inhibit elongation kinetics. In some cases, the cell has evolved intricate machinery to promote faster translation kinetics (eIF5A, for example) or to permit ribosome frameshifting to resume proper translation, but in other cases, the cellular mechanism that resolves these kinetic defects remains unknown. For instance, the discovery of specific tRNA-codon pairs that inhibit translation elongation remains quite puzzling. What about these particular pairs elicits a translation arrest by the ribosome? Why does the order of the tRNA-codon pair matter? Moving forward, it will be important to improve our understanding of the communication between the ribosomal P and A sites during elongation, as mediated by the codons, tRNAs and active site of the ribosome. Additionally, the role of the ribosomal E site remains to be fully defined. The general stimulatory factor eIF5A binds the unoccupied ribosome E site to stimulate peptide-bond formation when peptidyl-transfer kinetics are slow between the P-site and A-site tRNAs. Thus, the occupancy of the E site is likely to be a key indicator of the translation elongation rate at a particular site in the coding sequence. Further investigation into the coordination of the E, P and A sites using careful biochemical or single-molecule approaches should provide a deeper understanding of the dynamics of ribosome pauses during translation elongation and their influence on gene expression.
Translation termination (Fig. 3) is also a rich area for continued exploration. What are the determinants of stop codon recognition beyond the codons themselves? Recent studies in organisms that use termination codons for both coding and termination functions (sense and nonsense) increased the interest in models where termination is guided by the mRNP context near the termination codon. One potential candidate for directly stimulating termination is PABP120. However, although we have begun to characterize the diverse collection of mRNA-binding proteins using computational and biochemical approaches127, we still have little idea of the composition of a single mRNP and how this composition varies on mRNAs encoding different genes (or even the same gene). More relevant to our interests here, we have no idea how this mRNP context might influence the actions of the ribosome and translation factors. A more thorough investigation using both proteomic and single-molecule approaches to define an mRNP code will provide insight into the communication between the ribosome, translation factors and mRNPs as they relate to translation.
Finally, although the last phase of translation, ribosome recycling (Fig. 4), is reasonably well determined biochemically and structurally, potential connections between recycling and translation initiation through ABCE1 remain particularly intriguing. Although there is some evidence to suggest a role for ABCE1 in connecting these phases of translation, how ABCE1 works to recruit initiation factors and promote preinitiation complex assembly remains to be determined. Additionally, further dissection of the connections between the activity of recycling factors, the availability of translation-competent ribosomes and gene expression patterns (that is, how ribosome concentration affects gene-specific expression) will be of great importance to better understand the aetiology of ribosomopathies. Given all these areas of further investigation and the constant discovery of new regulatory factors and sequence elements that modulate translation, the field will continue to be an exciting area for scientific exploration.
|
 Fig. 1: Overview of eukaryotic translation.
Fig. 1: Overview of eukaryotic translation.  Fig. 2: Translation elongation and resolution of ribosome stalling.
Fig. 2: Translation elongation and resolution of ribosome stalling.  Fig. 3: Translation termination and the role of mRNP context.
Fig. 3: Translation termination and the role of mRNP context. 
 Fig. 4: Ribosome recycling and rescue.
Fig. 4: Ribosome recycling and rescue.