Имеется, однако, проблема с интерпретацией аналогичного 'сдвига' для Alu субклассов, когда субклассы определяются исходя из взаимной дивергенции скорее, чем характеристик подсемейства, особенно если CpG сайты не исключены [30]. Как показано на Рис. 2, снижение в среднем содержания CpG на Alu элемент на хромосоме Y довольно быстрое в молодом AluY подсемействе, но замедляется со временем и остается почти постоянным в старых Alu элементах. Напротив, все подсемейства Alu на Х хромосоме сохраняют грубо одну и ту же среднюю пропорцию CpG относительно аутосом. Др. словами, скорость распада CpG м. отличаться между более молодыми и более старыми подсемействами, создавая впечатление различий в скорости 'исчезновения'.
Ускоренная отцовская элиминация Alu элементов, описанная выше и 'сдвиг' в направлении GC-богатых Alu кластеров скорее всего являются проявлениями одного и того же отцовского процесса делетирования. Если аналогичный процесс происходит и у грызунов, то он д. протекать значительно быстрее, чем у людей. Изучение B1 и B2 SINEs не выявило 'сдвига' в направлении SINE кластеров, т.к. даже самые молодые элементы присутствовали в SINE-dense кластерах и не встречаются молодые SINEs на хромосоме Y [27]. На базе Alu модели м. предсказать, что самые молодые SINEs, которые локализуются вне SINE кластеров, уже элиминировались или мутировали у грызунов, особенно на Y хромосоме. Это ускоренное 'удаление' м.б. связано с более коротким репродуктивным циклом у грызунов, чем у людей. Альтернативное объяснение базируется на недавних изменениях
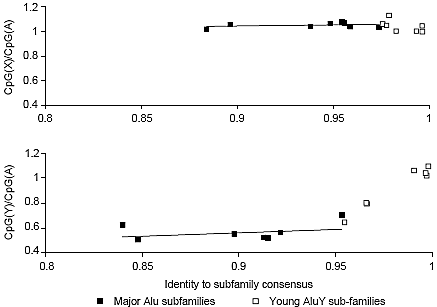 Proportions of CpG dinucleotide counts in Alu elements located on sex chromosomes and autosomes. Vertical axes show the ratios of the geometric mean of CpG counts per full-length Alu element on chromosome X (top) and chromosome Y (bottom), relative to those on autosomes. Horizontal
axes show average Alu element sequence identities to the consensus sequence of their corresponding subfamily. The major Alu subfamilies are Jo, Jb, Sq, Sx, Sg, Sc, Sp and Y (filled squares), and the young sub-subfamilies are Ya1, Ya4, Ya5, Yb8, Yc1, Yd2 and Ye5 (open squares). For details of Alu classification, see [20]. The different rates of CpG decay may reflect differences in methylation patterns between female and male germ lines [49].
Proportions of CpG dinucleotide counts in Alu elements located on sex chromosomes and autosomes. Vertical axes show the ratios of the geometric mean of CpG counts per full-length Alu element on chromosome X (top) and chromosome Y (bottom), relative to those on autosomes. Horizontal
axes show average Alu element sequence identities to the consensus sequence of their corresponding subfamily. The major Alu subfamilies are Jo, Jb, Sq, Sx, Sg, Sc, Sp and Y (filled squares), and the young sub-subfamilies are Ya1, Ya4, Ya5, Yb8, Yc1, Yd2 and Ye5 (open squares). For details of Alu classification, see [20]. The different rates of CpG decay may reflect differences in methylation patterns between female and male germ lines [49].
в механизмах интеграции SINE у людей по сравнению с грызунами [31,32]. Элиминация недавно вставленных SINEs м.б. обусловлена отбором против их повреждающих эффектов или неравных обменов (эктопических рекомбинаций)между разными SINE элементами во время митоза [33-37]. Др. процесс, такой как replication slippage между Alu элементами [38] также м. объяснять потерю молодых Alu элементов во всех хромосомах, включая Y хромосому, проходящих через отцовскую зародышевую линию.
Alu-mediated gene duplication,
rearrangements and alternative splicing
Alu дупликации более часты в Alu-богатых кластерах, чем в бедных Alu регионах хромосом [21]. Это м. отражать неравные обмены между кластерами Alu элементов, это м. приводить к делециям и дупликациям рекомбинирующих Alu элементов и хромосомных областей между ними. В отсутствие отбора количество делеций и удвоений д.б. одинаковым. В богатых генами областях, однако, удвоения ДНК скорее предоминируют над делециями, особенно если удвоенные сегменты длинные. Длинные, удвоенные хромосомные области обычно известны как low copy repeats (LCRs) или сегментные дупликации [39,40]. Они представляют от 5% до 6% генома человека [41] и имеют более высокое содержание GC, чем геном в среднем [21]. Однажды возникнув сегментные дупликации м. подвергаться дальнейшим не-аллельным гомологическим рекомбинациям [42] и Alu-обусловленным перестройкам [43], приводящим к генетическим болезням и эволюционным изменениям в геноме [44].
Модель участия Alu в возникновении сегментных дупликаций согласуется с недавно наблюдаемым обогащением субсемейств AluY и AluS вокруг соединений LCRs [45]. Эти два подсемейства размножились в последние 40 миллионов лет или около того, это согласуется с возрастом большинства LCRs. Не выявлено достоверного обогащения для более старых AluJ подсемейств. Обогащение Alu также более значительно внутри удвоенных сегментов, чем вне [45], это согласуется с моделью долговременного накопления Alu элементов в GC-богатых областях хромосом посредством удвоений Alu-обогащенных ДНК, возможно происходящих их LCRs [21].
В дополнение к роли Alu элементов в удвоениях и перестройках генов, Alu-обусловленный сплайсинг мРНК также м. приводить к новым мутациям и генетическим болезням, таким как Alport syndrome и gyrate atrophy of choroid или сетчатки [2]. Этот феномен был впервые описан более 10 лет тому назад [46], но до сих пор привлекает внимание [47,48] благодаря его потенциалу управлять эволюцией приматов путем генерации дополнительных вариантов белков при ограниченном количестве генов.
Conclusions
The propagation of Alu elements in the human genome
by L1-mediated retrotransposition triggers various genomic
mechanisms of Alu elimination and suppression. Such
mechanisms seem to be particularly effective in paternal
germ lines, where Alu retrotransposition seems to be most
active.
Alu-mediated unequal exchanges between meiotic chromosomes
can produce both deletions and duplications.
The latter are likely to survive in gene-rich chromosomal
regions where deletions would be harmful or lethal,
thereby stimulating evolution by gene duplication. Aside
from unequal exchanges, the elimination of Alu elements
may involve replication slippage fueled by higher rates of
germ-cell divisions in males than in females. In addition
to elimination, silencing by CpG methylation and accelerated
substitution rates are important for suppressing Alu
proliferation. Once fixed in the genome, Alu elements
may continue to produce alternatively spliced proteins,
which can be further tested by natural selection.
Studies of Alu elements significantly contribute to our
understanding of L1-mediated retrotransposition in
mammals and beyond. Current studies of genomic
response to Alu retrotransposition are expected to reveal
general mechanisms of post-insertion coevolution
between transposable elements and eukaryotic genomes.
Сайт создан в системе
uCoz Representation of female and male diploid cells that produce haploid
gametes. Top, female cells; bottom, male cells; A, X and Y indicate
autosomes and chromosomes X and Y, respectively. Numbers below
the chromosomes reflect average population weights of the
corresponding chromosomes. Arrows on the left and right side of
the male box indicate Alu insertions and Alu deletions, respectively.
On the basis of the paternal retrotranspositon model, the expected
proportions of the densities of newly inserted Alu elements on
chromosomes X and Y and autosomes are 1/3:1:1/2 or 1:3:3/2.
Analogous chromosomal proportions are expected for the rates of
replication-slippage-related Alu deletions in male germ lines.
Representation of female and male diploid cells that produce haploid
gametes. Top, female cells; bottom, male cells; A, X and Y indicate
autosomes and chromosomes X and Y, respectively. Numbers below
the chromosomes reflect average population weights of the
corresponding chromosomes. Arrows on the left and right side of
the male box indicate Alu insertions and Alu deletions, respectively.
On the basis of the paternal retrotranspositon model, the expected
proportions of the densities of newly inserted Alu elements on
chromosomes X and Y and autosomes are 1/3:1:1/2 or 1:3:3/2.
Analogous chromosomal proportions are expected for the rates of
replication-slippage-related Alu deletions in male germ lines.  Proportions of CpG dinucleotide counts in Alu elements located on sex chromosomes and autosomes. Vertical axes show the ratios of the geometric mean of CpG counts per full-length Alu element on chromosome X (top) and chromosome Y (bottom), relative to those on autosomes. Horizontal
axes show average Alu element sequence identities to the consensus sequence of their corresponding subfamily. The major Alu subfamilies are Jo, Jb, Sq, Sx, Sg, Sc, Sp and Y (filled squares), and the young sub-subfamilies are Ya1, Ya4, Ya5, Yb8, Yc1, Yd2 and Ye5 (open squares). For details of Alu classification, see [20]. The different rates of CpG decay may reflect differences in methylation patterns between female and male germ lines [49].
Proportions of CpG dinucleotide counts in Alu elements located on sex chromosomes and autosomes. Vertical axes show the ratios of the geometric mean of CpG counts per full-length Alu element on chromosome X (top) and chromosome Y (bottom), relative to those on autosomes. Horizontal
axes show average Alu element sequence identities to the consensus sequence of their corresponding subfamily. The major Alu subfamilies are Jo, Jb, Sq, Sx, Sg, Sc, Sp and Y (filled squares), and the young sub-subfamilies are Ya1, Ya4, Ya5, Yb8, Yc1, Yd2 and Ye5 (open squares). For details of Alu classification, see [20]. The different rates of CpG decay may reflect differences in methylation patterns between female and male germ lines [49].