Как только синтезируемая пре-мРНК оказывается capped, то ядерный связывающий шапочку белковый комплекс (cap-binding protein complex, CBC) соединяется ко-транскрипционно с монометилированной шапочкой. CBC состоит из CBP20, cap-binding protein и CBP80, которые гарантируют высокое сродство связывания и ассоциируют с CTD [16]. CBC, по-видимому, первый белок, который присоединяется к пре-мРНКs и необходим для последующих ступеней сплайсинга, decapping, трансляции и ядерного распада мРНК [17].
Установлено, что сплайсинг осуществляется по ходу транскрипции, хотя он м. происходить и отдельно от транскрипции [8,18]. CBC повышает эффективность связывания small nuclear RNP (snRNP) центральных компонентов spliceosome, таких как U1 snRNP, с cap-proximal 5' splice-сайтом, тем самым усиливается сборка предназначенного для сплайсинга комплекса на cap-proximal интроне. Осуществляемая по всему геному chromatin immunopreciptation (ChIP) предоставила доказательства ко-транскрипционного рекрутирования дрожжевого U1 snRNP, это было выявлено, особенно в интрон-содержащих генах [19]. Сплайсинг вносит вклад в формирование mRNPs из интрон-содержащих генов путём катализа отложения ~335 kDa exon-junction protein complex (EJC) 20-24 nt выше соединений между экзонами (Figure 2). У метазоа EJC содержат RNPS1, Y14, SRm160,
REF1/Aly, Upf3 и magoh белки и служат для маркировки РНК для нижестоящий ступеней процессинга, таких как экспорт, трансляция и nonsense-mediated decay (NMD). RNPS1, Y14, SRm160 и REF1/Aly обнаруживаются в ядерной CBP80-связанной мРНК [16,20] и вместе с UAP56, magoh и U snRNPs оказываютс видимы с помощью конфокальной микроскопии в местах транскрипции [21].
Однако, TAP-p15 и Dbp5 экспортные факторы не выявляются в местах транскрипции. Сборка EJC, как было установлено, происходит за счёт последующих ступеней в системе лизатов целой клетки, Aly/REF, RNPS1 и SRm160 были обнаружены на пре-мРНК во время формирования spliceosome, Upf3 непосредственно после первой splicing реакции, a Y14 и magoh после exon-exon ligation [22].
The interplay between transcription and cotranscriptional
assembly of export-competent mRNPs
Синтезируемая мРНК во время транскрипции покрывается белками, которые гарантируют интеграцию РНК и управляют экспортом РНК и нижестоящими цитоплазматическими ступенями. Если сборка mRNP не происходит собственно, то мРНК не может быть экспортирована, приводя к фенотипу оставления в ядре и деградации. Возможно, что многие из известных эукариотичеких РНК-связывающих белков играют главную роль в биогенезе мРНП, что согласуется с их обилием в клетке (Table 1). Тем не менее, большие количества предсказываемых РНК-связывающих белков и их отличающаяся по всему геному экспрессия и профили локализации белков открывают возможность, что некоторые могут преимущественно действовать на специфические суб-популяции мРНК и выполняют дополнительную роль в пост-трансрипционной регуляции [23,24,25,26].
Имеются многочисленные общие факторы важные для биогенеза готовых к экспорту mRNPs, которые включают Npl3, комплекс THO, Sub2/UAP56 и Yra1/Aly
(Figure 3). Npl3 является главным дрожжевым SR-подобным челночным РНК-связывающим белком (Table 1), котрорый рекрутируется на появляющуюся пре-мРНК во время транскрипции и играет роль в интеграции и экспорте mRNP [27]. Npl3 также совместно очищается с snRNAs, tRNAs и rRNAs [28].
THO является комплексом, состоящим из 4-х субъединиц (Tho2, Hpr1, Mft1 и
Thp2), который ассоциирует с Sub2/UAP56 РНК-зависимой АТФазой и Yra1/Aly/REF1 РНК-связывающим белком дрожжевого TREX комплекса [29-31]. Tho2 и Hpr1 очищаются вместе с UAP56 и Aly у людей, но свободны от UAP56 и Aly у
Drosophila [26,31]. Не перекрывающиеся геномные профили экспрессии
Drosophila THO- и Sub2-истощенных клеток [25,26] и высокий уровень дрожжевого Sub2 относительно THO (Table 1) [31,32] согласуются с тем, что Sub2/UAP56 выполнят дополнительную функцию, независимую от комплекса THO. THO, Sub2 и Yra1 рекрутируются во время транскрипции на кодирующие области, частично перекрываемые RNAPII, тогда как они не не связываются с неактивными генами, промоторами или транскрибируемыми регионами, стоящими ниже сайтов полиаденилирования [31,33,34]. Интерсно, что связывание поперечными связями дрожжевого Sub2 является THO- и РНК-зависимым, тогда как рекрутирование THO зависит от РНК. Следовательно, THO д. облегчать загрузку на выходящую мРНК Sub2 и Yra1 [33,35] и SR-подобных челночных РНК-связывающих белков, таких как Gbp2 и Hrb1, которые рекрутируются
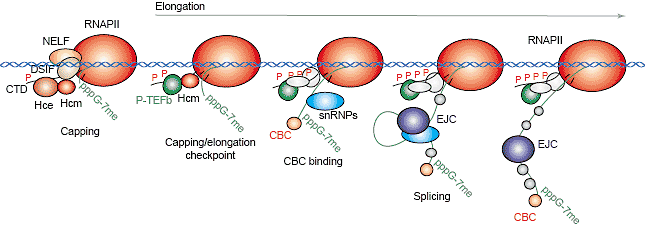 Schematic of the first steps of cotranscriptional mRNP assembly including 50-end capping, CBC (cap-binding protein complex) loading and
the splicing-dependent assembly of the exon junction complex (EJC), as it may occur in humans. After transcription initiation and phosphorylation
of the CTD-Ser2 of RNAPII by TFIIH-Cdk7, transcription is paused by the joint action of the DSIF and NELF transcription elongation factors,
allowing guanylyltransferase (Hce) and methyltransferase (Hcm) to cap the пре-мРНК 50-end in two steps as soon as it emerges from the
RNAPII. The capping machinery is displaced after completing its task, allowing the elongation factor P-TEFb to access the RNAPII CTD, which
leads to massive phosphorylation of Ser5 and to productive elongation being resumed. This serves as a checkpoint to prevent extension of
unproperly capped RNAs. Once the first intron is transcribed, the core splicing machinery of snRNPs is recruited to the intron and the EJC is
assembled onto the resulting exon-exon junction.
Schematic of the first steps of cotranscriptional mRNP assembly including 50-end capping, CBC (cap-binding protein complex) loading and
the splicing-dependent assembly of the exon junction complex (EJC), as it may occur in humans. After transcription initiation and phosphorylation
of the CTD-Ser2 of RNAPII by TFIIH-Cdk7, transcription is paused by the joint action of the DSIF and NELF transcription elongation factors,
allowing guanylyltransferase (Hce) and methyltransferase (Hcm) to cap the пре-мРНК 50-end in two steps as soon as it emerges from the
RNAPII. The capping machinery is displaced after completing its task, allowing the elongation factor P-TEFb to access the RNAPII CTD, which
leads to massive phosphorylation of Ser5 and to productive elongation being resumed. This serves as a checkpoint to prevent extension of
unproperly capped RNAs. Once the first intron is transcribed, the core splicing machinery of snRNPs is recruited to the intron and the EJC is
assembled onto the resulting exon-exon junction.
зависимым от транскрипции образом [36,37] (Figure 3). Chironomus и человеческий UAP56 также становится видимым в местах транскрипции [21,38].
THO мутанты обнаруживают связанные с transcription-elongation нарушения и зависимые от транскрипции гипер-рекомбинационные фенотипы [29,30,39] и быстрый распад мРНК [33,40]. Неэффективность транскрипционной элонгации у THO и sub2 мутантов подтверждена
in vitro и in vivo [41]. Более того, P Mason and K Struhl (personal
communication) показали с помощью ChIP, что RNAPII processivity
(нуклеотиды, синтезируемые на событие инициации) снижена у Spt4,5 и THO мутантов, это может объяснить неэффективность транскрипции и паттерны преждевременной утилизации вышестоящего poly-A сайта, наблюдаемые у этих мутантов [41,42,43].
THO действует во время транскрипционной элонгации, облегчаяя сборку готовых к экспорту mRNPs и предупреждая образование гибридов ДНК:РНК, это подтверждается наблюдениями, что hpr1D клетки ко-транскрипционно формируют ДНК:РНК гибриды и что их транскрипционные и рекомбинационные фенотипы зависят от длины сходящей (nascent) мРНК [3]. Это открывает неожиданную функцию для сборки mRNP: удерживание nascent РНК вдали от ДНК матрицы, предупреждая образование нежелательных структур, которые могут нарушить генетическую целостность (Figure 3). Обнаружение ДНК:РНК гибридов в class-switch регионах иммуноглобулинов, которые также участвуют в зависимой от транскрипции рекомбинации [44], ставит вопрос, может ли сборка mRNP играть роль в class switching.
В дополнение к ранее суммированным работам, др. экспериментальные доказательства подтверждают идею двухнаправленного взаимодействия между транскрипцией и формированием mRNP. Так, РНК-связывающий белковая изоформа hrp65-2 от
Chironomus ассоциирует с актином и влияет на транскрипционную элонгацию [45]. Elongator, законсервированный гистон
acetyl-transferase белковый комплекс (Elp1-6), который физически взаимодействует с elongating гипер-фосфорилированной RNAPII, связывает РНК и поперечно связывается
in vivo с RNAPII-производной пре-мРНК [46]. Субъединица NELF-E из NELF фактора элонгации человека является РНК связывающим белком [47]. Более того, функциональная карта кластеров разных элонгационных, сплайсирующих и экспортирующих компонентов показала, что дрожжевые Spt4-Spt5, Spt16, Spt6 и PAF транскрипционные факторы образуют кластер с THO, Sub2 и др. функциями биогенеза mRNP [48 ]. Наконец,
Drosophila U2AF50 сплайсинг фактор необходим для экспорта лишенных интронов мРНК [49], это подтверждает, что он выполняет более общую роль в биогенезе mRNP biogenesis. Интересно, что его человеческий ортолог U2AF65 ассоциирует с RNAPII прежде, чем будет загружен на транскрипт и оказывается необходимым для восстановления компетентности к транскрипционной элонгации в местах пауз (pause sites) [50].
Table 1
Abundance and localization of yeast proteins involved in mRNA biogenesis and metabolism and their respective metazoan orthologs.
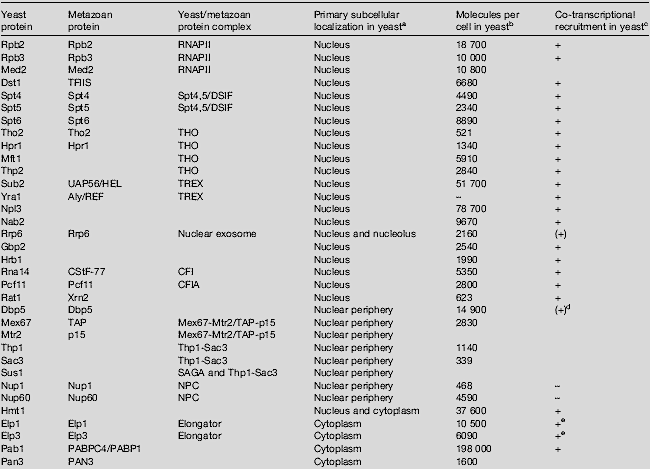 Blanks indicate that information is not known. aData taken from [70]. bData taken from [32]. cData obtained from different references as cited in the text. dIt has been shown for Chrironomus but there are no data regarding the yeast Dbp5. eShown to associate with RNAPII-driven
пре-мРНК.
Blanks indicate that information is not known. aData taken from [70]. bData taken from [32]. cData obtained from different references as cited in the text. dIt has been shown for Chrironomus but there are no data regarding the yeast Dbp5. eShown to associate with RNAPII-driven
пре-мРНК. Дальнейшее подтверждение важности сборки и процессинга mRNP, осуществляемых вместе с транскрипцией, получены благодаря наблюдению, что ядерные exosome ко-локализуются с факторами транскрипционной элонгации Spt5 т Spt6 в сайтах транскрипции у Drosophila [51,52]. Нарушения экспорта mRNP обусловливают сохранение РНК в сайтах транскрипции, где они предположительно деградируют зависимым от экзосом способом [17,40]. Более того, фенотипы нестабильности задержки в ядре РНК, наблюдаемые у THO транскрипционных termination/polyadenylation мутантов при высоких температурах, супрессируются с помощью общей редукции транскрипции, указывая тем самым, что частота транскрипции может модулироваться сборкой готовых к экспорту mRNPs [53]. Сходные результаты получены с мутантами
Dbp5/Rat8 РНК helicase, которые привели к предположению, что Dbp5/Rat8, который первично был локализован в ядерных порах (Table 1), может быть функционально связан с транскрипцией [54]. Правда, доказательства для ко-транскрипционного рекрутирования Dbp5 получены лишь у Chrironomus [1]. Возможно, что общее снижение транскрипции не приводит к предполагаемому коллапсу линии сборки и экспорта мРНК из-за того, что снижение общего уровня мРНК снижает спрос на heterogeneous nuclear RNPs (hnRNPs) и экспортные факторы. Однако, редукция эффективности транскрипции ингибирует также in vivo exon-skipping [55], супрессирует ssu72-2 мутации процессинга 3'-конца у дрожжей [56] и меняет альтернативный сплайсинг у Drosophila [57], указывая тем самым на точно регулируемое двунаправленное взаимодействие между формированием mRNP и транскрипцией.
Важность модификации белков в динамике сборки и процессинга mRNP начинает выявляться в исследованиях модификационных энзимов. Одним из таких энзимов является Hmt1, arginine methyl-transferase, участвующая в метилировании и транспорте ядро-цитоплазма мРНК-связывающих белков, таких как Nab2 [58]. Используя подходы к целому геному, в лаб. P Silver's была показана anHmt1- и РНК-зависимая ассоциация между Npl3 и Tho2. Профили связывания РНК с Hrp1 3'-концом
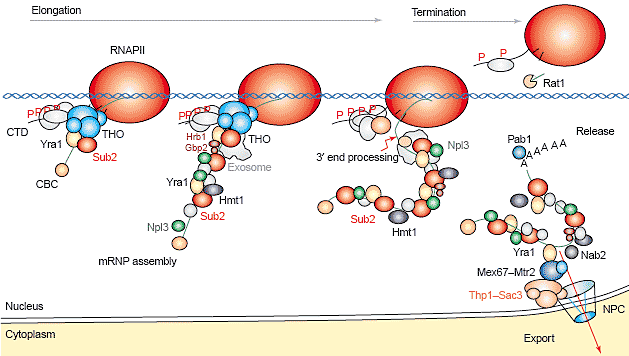 Simplified model of co-transcriptional mRNP assembly, as it might occur in the intron-less genes of the yeast S. cerevisiae. During transcription
elongation, the THO complex facilitates the loading of subunits of the Sub2 (metazoan UAP56) RNA-dependent ATPase and the Yra1 RNA
binding export factor (metazoan Aly/REF1) along the nascent mRNA molecule. This view is consistent with the high abundance of Sub2
(see Table 1) and is also supported by the facts that multicopy Sub2 suppresses THO mutants and that Sub2 recruitment is THO- and
RNA-dependent (see text). Many other proteins are assembled onto the nascent mRNA (Npl3, Hrb1, Gbp2) or work during co-transcriptional
assembly (Hmt1) to modulate protein-protein or protein-RNA interactions (see Table 1). As a result an export-competent mRNP is formed.
The mRNA 30-end is processed by the cleavage and polyadenylation factors that are recruited via the CTD. Once the mRNP is released from
the site of transcription by the action of the 50-30 exoribonuclease Rat1 (metazoan Xrn2), the mRNP is exported. The Mex67-Mtr2 export
factor (metazoan TAP-p15) binds Yra1, displacing Sub2 from Sub2-Yra1 heterodimers, to export the mRNP via other RNA binding factors
(Thp1-Sac3 complex, Nab2, etc). If mRNP is not properly assembled during transcription elongation, downstream steps such as transcript
termination, 30-end processing and release from the transcription site could be affected, and the mRNA would not be exported and would
undergo exosome-mediated degradation. In mutants of THO/TREX, in particular, the nascent mRNA would form DNA:RNA hybrids behind the
elongating RNAPII, triggering genetic instability and reducing RNAPII processivity.
Simplified model of co-transcriptional mRNP assembly, as it might occur in the intron-less genes of the yeast S. cerevisiae. During transcription
elongation, the THO complex facilitates the loading of subunits of the Sub2 (metazoan UAP56) RNA-dependent ATPase and the Yra1 RNA
binding export factor (metazoan Aly/REF1) along the nascent mRNA molecule. This view is consistent with the high abundance of Sub2
(see Table 1) and is also supported by the facts that multicopy Sub2 suppresses THO mutants and that Sub2 recruitment is THO- and
RNA-dependent (see text). Many other proteins are assembled onto the nascent mRNA (Npl3, Hrb1, Gbp2) or work during co-transcriptional
assembly (Hmt1) to modulate protein-protein or protein-RNA interactions (see Table 1). As a result an export-competent mRNP is formed.
The mRNA 30-end is processed by the cleavage and polyadenylation factors that are recruited via the CTD. Once the mRNP is released from
the site of transcription by the action of the 50-30 exoribonuclease Rat1 (metazoan Xrn2), the mRNP is exported. The Mex67-Mtr2 export
factor (metazoan TAP-p15) binds Yra1, displacing Sub2 from Sub2-Yra1 heterodimers, to export the mRNP via other RNA binding factors
(Thp1-Sac3 complex, Nab2, etc). If mRNP is not properly assembled during transcription elongation, downstream steps such as transcript
termination, 30-end processing and release from the transcription site could be affected, and the mRNA would not be exported and would
undergo exosome-mediated degradation. In mutants of THO/TREX, in particular, the nascent mRNA would form DNA:RNA hybrids behind the
elongating RNAPII, triggering genetic instability and reducing RNAPII processivity.
processing белка и Nab2 hnRNP, изменены в hmt1D клетках, указывая тем самым, что Hmt1 может предопределять архитектуру mRNP, модулируя межбелковые или РНК-белок взаимодействия [24]. Более того, экспортная функция Npl3 обеспечивается с помощью взаимодействия с фактором экспорта Mex67; это взаимодействие нуждается в Glc7 phosphatase-зависимом дефосфорилировании Npl3 [59 ]. Сходным образом. взаимодействие между адаптерными SR белками и TAP регулируется с помощью дефосфорилирования [60]. Предполагаемая важность модификаций белков для архитектуры и динамики mRNP ставит вопрос, могут ли активности, такие как РНК-зависимая ATФase из встречающегося в большом количестве белка
Sub2/UAP56 [61], также играть роль в становлении межбелковых и белок-РНК взаимодействий вдоль mRNPs.
От мест транскрипции через ядерные поры mRNPs экспортируется в цитоплазму после завершения транскрипции и высвобождения от сайта транскрипции. Это высвобождение связано с действием нескольких белков на только что синтезированную мРНК, включая обрезание polyA хвоста с помощью PAN poly-A nuclease и загрузку Pab1 на мРНК (у дрожжей) [62] и действие Rat1/Xrn2 5'-3' exonucleases (у дрожжей и людей) [63,64]. Особенно дрожжевые мутанты по транскрипционной элонгации, 3'-концевому процессингу и экспорту имеют перекрывающиеся фенотипы [13,48,65], указывая тем самым на интригующую взаимозависимость между сборкой готовых к экспорту mRNPs
и 3'-концевым процессингом и терминацией, которую необходимо понять, чтобы полностью расшифровать механизмы co-transcriptional образования mRNP.
Терминация транскрипции и 3'-концевой процессинг рассмотрены в параллельном обзоре Buratowski.
Наконец, новые находки показали, экспорт мРНК может быть облегчен её физической и субклеточной ко-локализацией с транскрипцией, а , следовательно, с co-transcriptional сборкой mRNP. В лаб. E Hurt's наблюдали, что новый дрожжевой белок, Sus1, работает в промоторных областях как часть SAGA histone-acetylase транскрипционного комплекса и в nuclear pore complexes (NPCs), как часть Thp1-Sac3 комплекса экспорта РНК [66], он также влияет на биогенез и рекомбинацию мРНК [67]. В лаб.
P Silver's было установлено, что факторы ядерного транспорта, nucleoporins и karyopherins ассоциируют с субнабором высоко транскрибируемых генов и что активация GAL генов приводит в результате к их клеточной ассоциации с NPCs. Это указывает на то, что активные сайты транскрипции субнабора генов могут быть локализованы в NPCs [68], это могло бы объяснить, почему мутации в THO, Sub2, Yra1, Nab2,
Mtr2-Mex67 или Thp1-Sac3 факторов, которые работают на разных стадиях конвейера биогенеза и экспорта mRNP (Table 1, Figure 3), дают сходные фенотипы биогенеза мРНК и геномной нестабильности
[39,65,68,69].
Conclusions and perspectives
We are beginning to realize the importance in mRNP
biogenesis of proteins relevant to the transcription cycle,
to RNA integrity and export and to genetic integrity. As
the number of proteins identified as co-transcriptionally
assembling onto the пре-мРНК increases, we have to
distinguish those with an architectural role from those
with a functional role (in export, decay, processing, etc) as
well as the putative protein modifications (methylation,
phosphorylation, etc) that modulate their interactions.
Furthermore, other questions are raised. Do cells have
specific transcription-coupled mRNP-assembly machinery?
Are there checkpoint mechanisms that limit the
production of improperly assembled mRNPs? Is there
a mechanistic interdependence between mRNP assembly
and RNAPII processivity? Can mRNP formation
control the expression of specific subsets of genes requiring
a fast response, such as stress-regulated genes? Does
mRNP assembly play a role, in addition to helping to
maintain general genome integrity, in the control of
developmentally regulated processes of recombination?
These and other questions will keep our interest on this
exciting field in the near future.
Update
The work of P Mason and K Struhl on the analysis of
transcription elongation rate and processivity in different
transcription and mRNP biogenesis factors is now in
press [71]. Also, additional data have been provided
on the negative impact on RNA synthesis of yeast yra1
mutants, which show that mRNP particles are sequestered
together with the perinuclear Mlp proteins [72].
These reports provide further evidence for coupling
between transcription and assembly of export-competent
mRNPs.
Сайт создан в системе
uCoz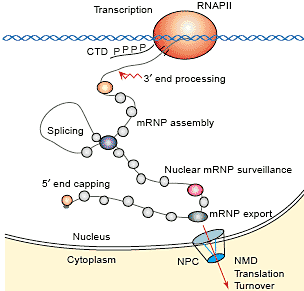 Simplified view of eukaryotic gene expression, covering transcription
and the nuclear mRNP processing steps of 50-end capping, splicing,
mRNP assembly, 30-end cleavage and polyadenylation, mRNP
surveillance and RNA export, which are coupled or coordinated with
transcription. NMD, nonsense-mediated decay; NPC, nuclear pore
complex; RNAPII, RNA polymerase II.
Simplified view of eukaryotic gene expression, covering transcription
and the nuclear mRNP processing steps of 50-end capping, splicing,
mRNP assembly, 30-end cleavage and polyadenylation, mRNP
surveillance and RNA export, which are coupled or coordinated with
transcription. NMD, nonsense-mediated decay; NPC, nuclear pore
complex; RNAPII, RNA polymerase II.  Schematic of the first steps of cotranscriptional mRNP assembly including 50-end capping, CBC (cap-binding protein complex) loading and
the splicing-dependent assembly of the exon junction complex (EJC), as it may occur in humans. After transcription initiation and phosphorylation
of the CTD-Ser2 of RNAPII by TFIIH-Cdk7, transcription is paused by the joint action of the DSIF and NELF transcription elongation factors,
allowing guanylyltransferase (Hce) and methyltransferase (Hcm) to cap the пре-мРНК 50-end in two steps as soon as it emerges from the
RNAPII. The capping machinery is displaced after completing its task, allowing the elongation factor P-TEFb to access the RNAPII CTD, which
leads to massive phosphorylation of Ser5 and to productive elongation being resumed. This serves as a checkpoint to prevent extension of
unproperly capped RNAs. Once the first intron is transcribed, the core splicing machinery of snRNPs is recruited to the intron and the EJC is
assembled onto the resulting exon-exon junction.
Schematic of the first steps of cotranscriptional mRNP assembly including 50-end capping, CBC (cap-binding protein complex) loading and
the splicing-dependent assembly of the exon junction complex (EJC), as it may occur in humans. After transcription initiation and phosphorylation
of the CTD-Ser2 of RNAPII by TFIIH-Cdk7, transcription is paused by the joint action of the DSIF and NELF transcription elongation factors,
allowing guanylyltransferase (Hce) and methyltransferase (Hcm) to cap the пре-мРНК 50-end in two steps as soon as it emerges from the
RNAPII. The capping machinery is displaced after completing its task, allowing the elongation factor P-TEFb to access the RNAPII CTD, which
leads to massive phosphorylation of Ser5 and to productive elongation being resumed. This serves as a checkpoint to prevent extension of
unproperly capped RNAs. Once the first intron is transcribed, the core splicing machinery of snRNPs is recruited to the intron and the EJC is
assembled onto the resulting exon-exon junction.  Blanks indicate that information is not known. aData taken from [70]. bData taken from [32]. cData obtained from different references as cited in the text. dIt has been shown for Chrironomus but there are no data regarding the yeast Dbp5. eShown to associate with RNAPII-driven
пре-мРНК.
Blanks indicate that information is not known. aData taken from [70]. bData taken from [32]. cData obtained from different references as cited in the text. dIt has been shown for Chrironomus but there are no data regarding the yeast Dbp5. eShown to associate with RNAPII-driven
пре-мРНК.  Simplified model of co-transcriptional mRNP assembly, as it might occur in the intron-less genes of the yeast S. cerevisiae. During transcription
elongation, the THO complex facilitates the loading of subunits of the Sub2 (metazoan UAP56) RNA-dependent ATPase and the Yra1 RNA
binding export factor (metazoan Aly/REF1) along the nascent mRNA molecule. This view is consistent with the high abundance of Sub2
(see Table 1) and is also supported by the facts that multicopy Sub2 suppresses THO mutants and that Sub2 recruitment is THO- and
RNA-dependent (see text). Many other proteins are assembled onto the nascent mRNA (Npl3, Hrb1, Gbp2) or work during co-transcriptional
assembly (Hmt1) to modulate protein-protein or protein-RNA interactions (see Table 1). As a result an export-competent mRNP is formed.
The mRNA 30-end is processed by the cleavage and polyadenylation factors that are recruited via the CTD. Once the mRNP is released from
the site of transcription by the action of the 50-30 exoribonuclease Rat1 (metazoan Xrn2), the mRNP is exported. The Mex67-Mtr2 export
factor (metazoan TAP-p15) binds Yra1, displacing Sub2 from Sub2-Yra1 heterodimers, to export the mRNP via other RNA binding factors
(Thp1-Sac3 complex, Nab2, etc). If mRNP is not properly assembled during transcription elongation, downstream steps such as transcript
termination, 30-end processing and release from the transcription site could be affected, and the mRNA would not be exported and would
undergo exosome-mediated degradation. In mutants of THO/TREX, in particular, the nascent mRNA would form DNA:RNA hybrids behind the
elongating RNAPII, triggering genetic instability and reducing RNAPII processivity.
Simplified model of co-transcriptional mRNP assembly, as it might occur in the intron-less genes of the yeast S. cerevisiae. During transcription
elongation, the THO complex facilitates the loading of subunits of the Sub2 (metazoan UAP56) RNA-dependent ATPase and the Yra1 RNA
binding export factor (metazoan Aly/REF1) along the nascent mRNA molecule. This view is consistent with the high abundance of Sub2
(see Table 1) and is also supported by the facts that multicopy Sub2 suppresses THO mutants and that Sub2 recruitment is THO- and
RNA-dependent (see text). Many other proteins are assembled onto the nascent mRNA (Npl3, Hrb1, Gbp2) or work during co-transcriptional
assembly (Hmt1) to modulate protein-protein or protein-RNA interactions (see Table 1). As a result an export-competent mRNP is formed.
The mRNA 30-end is processed by the cleavage and polyadenylation factors that are recruited via the CTD. Once the mRNP is released from
the site of transcription by the action of the 50-30 exoribonuclease Rat1 (metazoan Xrn2), the mRNP is exported. The Mex67-Mtr2 export
factor (metazoan TAP-p15) binds Yra1, displacing Sub2 from Sub2-Yra1 heterodimers, to export the mRNP via other RNA binding factors
(Thp1-Sac3 complex, Nab2, etc). If mRNP is not properly assembled during transcription elongation, downstream steps such as transcript
termination, 30-end processing and release from the transcription site could be affected, and the mRNA would not be exported and would
undergo exosome-mediated degradation. In mutants of THO/TREX, in particular, the nascent mRNA would form DNA:RNA hybrids behind the
elongating RNAPII, triggering genetic instability and reducing RNAPII processivity.