Endoplasmic Reticulum–Associated Degradation | |
|
| Секреторные и трансмембранные белки вступают на секреторны путь посредством белок-проводящего Sec61 канала в мембране эндоплазматического ретикулема. В эндоплазматическом ретикулеме, белковая укладка часто ковалентно модифицируется и олигомеризуется прежде чем быть упакованы в транспортные пузцрьки, которые доставят их в комплекс Гольджи. Белки, которые оказались упакованы неправильно в эндоплазматическом ретикулеме избирательно транспортируются назад через мембрану эндоплазматического ретикулема в цитозоль для деградации с помощью протеосом. В зависимости от топологии дефекта в белке, цитозольные или просветные хапероны (chaperones) вовлекаются в из отправку на деградацию. Экспортным каналом для неправильно уложенных белков также является Sec61p. Экспорт может подкрепляться с помощью AAA-ATPases из 19S регуляторной частицы протеасом или с помощью Cdc48p/p97. Экспортируемые белки часто ubiquitylated
перед деградацией и экскортируются на протеосомы с помощью polyubiquitin-связывающих белков.
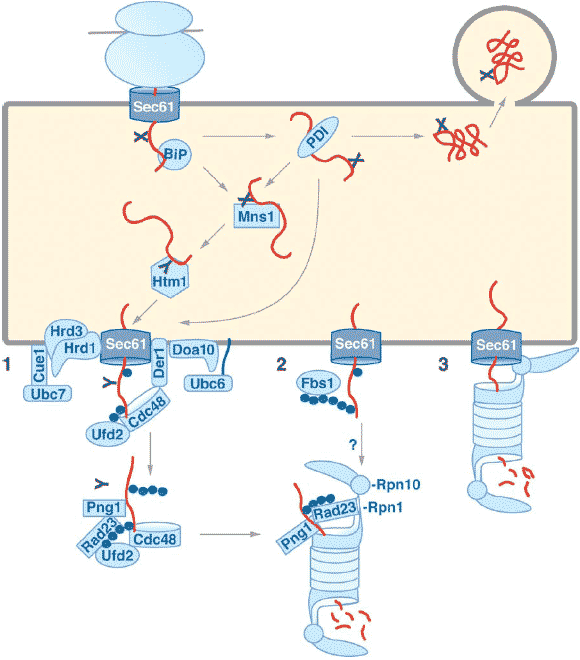 Figure 1
ER-associated degradation. Secretory proteins enter the ER through the Sec61 channel cotranslationally (depicted here) or posttranslationally. Proteins carrying appropriate acceptor sites are
N-glycosylated (black X). Proteins fold with the help of lumenal chaperones such as BiP and PDI. Fully folded proteins are packaged into ER-to-Golgi transport vesicles. Misfolding leads to prolonged
association with chaperones, trimming of a mannose residue from N-glycans by ER-mannosidase I (Mns1p) and recognition of the trimmed N-glycan (black Y ) by Htm1p, which targets misfolded glycoproteins for degradation. Targeting for export of nonglycosylated proteins may be mediated
directly by PDI. Misfolded proteins are exported to the cytosol through a channel most likely formed by Sec61p and multiple accessory proteins. During export, misfolded proteins are oligo-ubiquitylated (blue
circles) by either Hrd1p (E3) and Ubc7p (E2) or Doa10p (E3) and Ubc6p (E2). (1) Der1p-anchored Cdc48p at the ER membrane may recognize oligo-ubiquitylated substrates and assist their size-restricted polyubiquitylation by Ufd2p (E4). After substrate deglycosylation by Png1p, Rad23p (or Dsk2p) can bind to the Ufd2p-associated polyubiquitin and target the substrate to the proteasome by binding the 19S RP subunit Rpn1p. Some or all of the factors involved may be bound to the ER
membrane. (2) Alternatively, polyubiquitylation may be mediated by the N-glycan-specific SCFFbs1 complex; the resulting longer polyubiquitin chains may be recognized by the 19S RP subunit
Rpn10p. (3) In a third scenario, the 19S RP binds directly to the Sec61 channel and extracts misfolded proteins from the ER. If 20S core particles associate with these 19S RP complexes, as shown here,
export and degradation can be coupled. Alternatively, 19S RPs could function on their own in driving export similar to Cdc48p in scenario (1). In this case, some or all of the steps depicted in (1) and (2) may
also precede proteolysis when the driving force for export is provided by the 19S RP. Figure 1
ER-associated degradation. Secretory proteins enter the ER through the Sec61 channel cotranslationally (depicted here) or posttranslationally. Proteins carrying appropriate acceptor sites are
N-glycosylated (black X). Proteins fold with the help of lumenal chaperones such as BiP and PDI. Fully folded proteins are packaged into ER-to-Golgi transport vesicles. Misfolding leads to prolonged
association with chaperones, trimming of a mannose residue from N-glycans by ER-mannosidase I (Mns1p) and recognition of the trimmed N-glycan (black Y ) by Htm1p, which targets misfolded glycoproteins for degradation. Targeting for export of nonglycosylated proteins may be mediated
directly by PDI. Misfolded proteins are exported to the cytosol through a channel most likely formed by Sec61p and multiple accessory proteins. During export, misfolded proteins are oligo-ubiquitylated (blue
circles) by either Hrd1p (E3) and Ubc7p (E2) or Doa10p (E3) and Ubc6p (E2). (1) Der1p-anchored Cdc48p at the ER membrane may recognize oligo-ubiquitylated substrates and assist their size-restricted polyubiquitylation by Ufd2p (E4). After substrate deglycosylation by Png1p, Rad23p (or Dsk2p) can bind to the Ufd2p-associated polyubiquitin and target the substrate to the proteasome by binding the 19S RP subunit Rpn1p. Some or all of the factors involved may be bound to the ER
membrane. (2) Alternatively, polyubiquitylation may be mediated by the N-glycan-specific SCFFbs1 complex; the resulting longer polyubiquitin chains may be recognized by the 19S RP subunit
Rpn10p. (3) In a third scenario, the 19S RP binds directly to the Sec61 channel and extracts misfolded proteins from the ER. If 20S core particles associate with these 19S RP complexes, as shown here,
export and degradation can be coupled. Alternatively, 19S RPs could function on their own in driving export similar to Cdc48p in scenario (1). In this case, some or all of the steps depicted in (1) and (2) may
also precede proteolysis when the driving force for export is provided by the 19S RP. |