An hypothesis about genome structures in mammalian polyploid cells based on a new concept that genome is fractal of six huerarchies | |
|
Согласно новой концепции структура ДНК является фракталем с единицами из двойных окружностей с противоположной направленностью. В диплоидных клетках человека ДНК сконструирована из 6 иерархий, чьими размерами являются 325, 324, 323, 322, 321 и 320 с единицей в 200 п.н. ДНК, соответствующих генеому, хромосоме, хромосомному диску, репликону, гену и нуклеосоме, соответственно. Новая концепция удовлетворительно объясняет проблему потери ДНК в полиплоидных клетках. Гомологичные хромосомы полиплоидных клеток располагаются зеркально симметрично и они могут приходить в тесное соприкосновение, образуя скадчатую структуру. Когда синтез ДНК обходит спаренную гомологичную хромосому, то содержимое ДНК теряется при каждом делении.
|
ДНК содержимое полиплоидных клеток иногда снижается постепенно и случано внезапно снижается вдвое. Проведены некоторые исследования уменьшения плоидности клеток. Moor et al., [1] Пришли к заклЮчениЮ, что почти триплоидность является терминальным состоянием почти тетраплоидности в клеткаХ асцитической опуХоли Ehrlich. Harris [2] показал, что число хромосом константно в диплоидных клетках, но снижается при субкультивировании тетраплидных и октаплоидных клеток почек свиней. Graves, McMillan [3] сообщили, что тераплоидные DON и Bl клетки кит. хомячка кариотивически стабильны, но октаплидные клетки нет.
Несмотря на эти долговременные исследования механизм уменьшения ДНК неизвестен. Первоначальная концепция заключалась в том, что хромосомы, располагаясь независимо в ядре, не могут объяснить этих результатов.
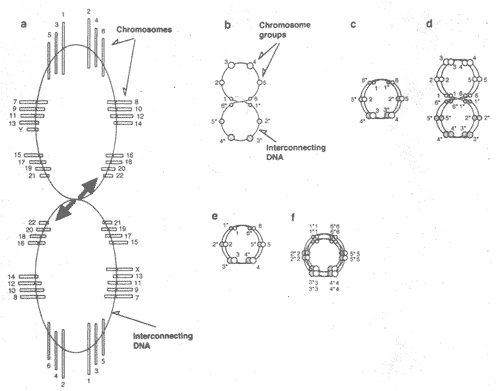 Figure 1 A model for genome structures in cells. All of the chromosomes are connected at centromeres by a double-strand DNA, interconnecting DNA. In somatic cells, homologous chromosomes are point-symmetrically located so that larger chromosomes are situated far apart from each other and the smaller ones are positioned nearer each other (a). Note that the exact location and order of chromosome are not known. The configuration shown in (a) is schematically drawn in (b). Small circles 1 and 6, 2 and 5, and 3 and 4 represent sets of short, medium-sized and long chromosomes, respectively. One to 6 and 1* to 6* represent paternal and maternal haploid sets, respectively. In the folded structure, homologous chromosomes cannot be close to each other (c). DNA synthesis produces genome pairs that cannot be separated without strand breakage (d). In germ cells, by unknown mechanisms such as pulling between sex chromosomes, all of the homologous chromosomes are allowed to be in close proximity in the folded structure (e). DNA synthesis produces tetramers of homologous chromosomes (f). The arrows indicate the direction of signals.
Figure 1 A model for genome structures in cells. All of the chromosomes are connected at centromeres by a double-strand DNA, interconnecting DNA. In somatic cells, homologous chromosomes are point-symmetrically located so that larger chromosomes are situated far apart from each other and the smaller ones are positioned nearer each other (a). Note that the exact location and order of chromosome are not known. The configuration shown in (a) is schematically drawn in (b). Small circles 1 and 6, 2 and 5, and 3 and 4 represent sets of short, medium-sized and long chromosomes, respectively. One to 6 and 1* to 6* represent paternal and maternal haploid sets, respectively. In the folded structure, homologous chromosomes cannot be close to each other (c). DNA synthesis produces genome pairs that cannot be separated without strand breakage (d). In germ cells, by unknown mechanisms such as pulling between sex chromosomes, all of the homologous chromosomes are allowed to be in close proximity in the folded structure (e). DNA synthesis produces tetramers of homologous chromosomes (f). The arrows indicate the direction of signals.
A model for spatial structures of DNA in diploid cells Genome structures deduced from observations
Имеется множество наблюдений о структуре генома, которые игнорируются. Myhra, Brogger [4] продемонстрировали, что хромосомы соединены по центромерам с помощью межхромосомных нитей ДНК. Wollenberg et al., [5] изучали пространственное взаимоотношение между гомологичными парами хромосом в нормальных клетках человека и пришли к выводу, базирующемуся на статистических выкладках, что хромосомы располагаются так, что более крупные хромосомы находтся от любой др. как можно дальше, а более мелкие позиционируются вблизи др. др. Nagele et al., [6] показали. что среднего размера гомологичные хромосомы располагаются на оппозитных сторонах хромосомной розетки.
Эти наблюдения подтверждают модель структуры генома в клетках, показанную на Рис. 1. Согласно этой модели все хромосомы сцеплены по центромерам с помощью двунитчатой ДНК, обозначаемой как взаимосоединяющая (interconnecting) ДНК. В соматических клетках генома хромосомы расположены pion-symmetrically, располагаясь так, что наиболее крупные хромсомы находятся дальше от всех др., а наиболее мелкие располагаются вблизи др. др.(Рис. 1 (а) и (b)), В сложенной структуре гомологичные хромосомы не могт придти в тесное воприкосновение др. с др. в соматических клетках (Рис. 1 (с), но в зародышевых клетках они делают это (Рис. 1 (е)). Специфические конфигурации гомологичных хромосом м. обеспечивать в результате удивительную стабильность содержимого ДНК в соматических диплоидных клетках млекопитающих. С др. стороны, парные конфигурации гомологичных хромосом в сложенной (folded) структуре зародышевых клеток делают возможными синапсы всех гомологичных хромосом в мейозе без каких-либо затруднений, связанных с вероятностями спаривания (Рис. 1 (f)). Точная локализация хромосом всё ещё неизвестна, однако, последовательность разделения центросом описана [7,8]. Пока не proved, что хромосомы независимы, хотя эмбпирически это распозанется.
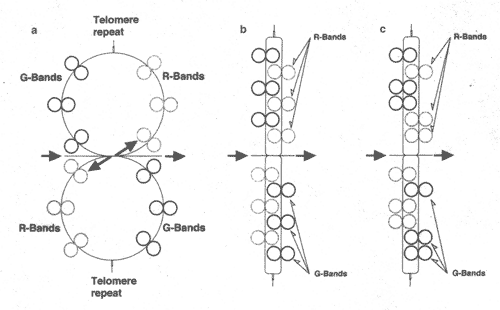 Figure 2 A model for chromosome structures in cells. Chromosomes are twin-loop configurations and consist of R-bands, G-bands and interconnecting DNA that connects the bands (a). Signals such as condensation and decondensation signals are transmitted along the interconnecting DNA, as indicated by arrows. When DNA-strands are coiled, chromosomes become rod shaped. Telomeres are recognized as a useful structure to relieve the stress in coiled DNA-strands and to prevent strand breaks (b). Shortening of telomeres leads to strand breakage and incomplete DNA • winding and results in cell death. As the coiling in the DNA strand progresses, adjacent bands in the structure will combine and become a larger band (c).
Figure 2 A model for chromosome structures in cells. Chromosomes are twin-loop configurations and consist of R-bands, G-bands and interconnecting DNA that connects the bands (a). Signals such as condensation and decondensation signals are transmitted along the interconnecting DNA, as indicated by arrows. When DNA-strands are coiled, chromosomes become rod shaped. Telomeres are recognized as a useful structure to relieve the stress in coiled DNA-strands and to prevent strand breaks (b). Shortening of telomeres leads to strand breakage and incomplete DNA • winding and results in cell death. As the coiling in the DNA strand progresses, adjacent bands in the structure will combine and become a larger band (c).
Chromosome structures deduced from observations Имеется множество наблюдений, проливающих свет на присущую картину структур хромосом. Bak et al., [9] получили фотографии кольцеобразных хромосом в клетках человека. Кольце-образные структуры хромосом описаны [10-12]. Очевидно, что хромосомы имеют кольце-образную структуру. Предполагается, что хромосомы млекопитающих приобретают колцевидную структуру в аномальных ситуациях, таких как рак, однако нет прямых доказательств, что хромосомы имеют линейную форму в номральных клетках.
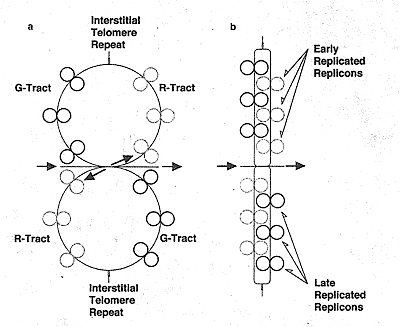 Figure 3 A structural model for chromosome bands in chromosomes. Chromosome bands are twin-loop configurations that consist of early-replicating replicons in the R-tracts, late-replicating replicons in the G-tracts and interconnecting DHA that connects replicons (a). Signals such as condensation and decondensation signals are transmitted along interconnecting DNA, as indicated by arrows. When DNA-strands are coiled, chromosome bands may become rod shaped. Interstitial-telomeres are recognized as useful structures to relieve the stress in coiled DNA-strands and to prevent strand breaks (b).
Figure 3 A structural model for chromosome bands in chromosomes. Chromosome bands are twin-loop configurations that consist of early-replicating replicons in the R-tracts, late-replicating replicons in the G-tracts and interconnecting DHA that connects replicons (a). Signals such as condensation and decondensation signals are transmitted along interconnecting DNA, as indicated by arrows. When DNA-strands are coiled, chromosome bands may become rod shaped. Interstitial-telomeres are recognized as useful structures to relieve the stress in coiled DNA-strands and to prevent strand breaks (b). Хромосомные диски уменьшаются в числе с G2 до М фазы. Согласно т.наз. правилу трех дисков, один минорный субдиск окружен двумя основными субдисками, слитыми высоко скоординированным способом [13]. Согласно правилу Ganner-Evance R-диски содерждат гены домашнего хозяйства и реплицируются прежде G-дисков [14]. Эти два правила могут подтверждать кольцеобразную структуру хромосом, как показано на Рис. 2. В этой модели хромосом состоят из двух регионов; R- и G-областей, где R- и G-диски, соотв. связаны с помощью interconnecting ДНК.
В этой модели interconnecting ДНК играет важную роль, функционируя как передатчик сигналов от хромосомного диска к хромосомному диску, подобно fuse-code. Сигналы, такие как де-конденсирующие и конденсирующие сигналы, циркулируют от R- к G- областям и touch off начинают передавать сигналы к хромосомным дискам (Рис. 2(а)). Если хромосомы принимают структуры двойной петли, то теломеры д. служить как действующие структуры, чтобы воспринимать стрессы в скрученных нитях ДНК и предупредить разрывы нити. Conceivable structures of chromosome bands based on observations
Теломеры содержат специфические посторы оснований ДНК (TTAGGG)n , еоторые называются теломерными повторами. Многие теломерные посторы обнаруживаются в областях, иных, чем теломеры и они наз. интерстициальными теломерными повторами [15]. Возможно, что роль теломерных повторов идентична у теломер и интерстициальных теломер. Очень длинная цепь двунитчатой ДНК, связаннfz водородными мостиками, может пережить стресс в скрученных ДНК нитях и предупридить разрывы нитей. Присутствие интерстициальных теломерных повторов подтверждает структуру хромосомных дисков, сходную со структурой хромосом, как показано на Рис. 3. В этой модели хромосомный диск конструируется двумя треками, R- и G-треками , в которых находятся рано- и поздно-реплицирующиеся репликоны, соотв., связанные соединительной ДНК.
Fractal structures of DNA in human cells Фрактальная геометрия широко используется, чтобы охарактеизовать структуры, включающие похожие (self-similarity) формы [16]. Если предположить self-similarity в форме структур генома в виде фракталя со структурными единицами противоположной ручности (handedness) из кольцевых пар, то репликоны д.б. представлены много меньшими и сходными структурами ДНК. Это может быть розетка петель, которая может содержать по одному гену на петлю. В клетках человека средний размер ДНК генома, хромосомы, хромосомного диска, репликона, розетки петель и нуклеосомы примерно равен 6х109, 1.3х108, 3х106, 1.2х105, 5х103 и 2х102 оснований, соотв. Соотношение этих размеров может быть грубо представлено как 325:324:323:321:320.возможно, что структура ДНК в клетках человека является фрактальной из 6 иерархий с само-похожестью форм в виде структурной единицы из пары окружностей с противопложрной ручностью.
A model for genome structures of polyploid cells 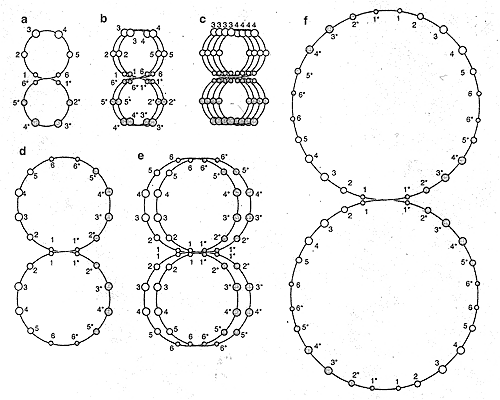 Figure 4 Models of the DNA structure for diploid (a), tetraploidlzed (b), tetraploid (d), octaploidized (c, e) and octaploid(f) cells. In diploid cells, DMA is connected as homologous chromosomes arranged with point symmetry. Small circles 1 and 6, 2 and 5, and 3 and 4 represent sets of short, medium-sized and long chromosomes, respectively. One to 6 (open circles) and 1* to 6* (closed circles) represent paternal and maternal haploid sets, respectively. When DNA is synthesized without cell division, the DNA twin circles are multiplied, resulting in tetraploidized (b) and octaploidized (c, e) configurations of DNA. Note that the DNA configuration of octaploidized cells is different from the configuration In diploid and tetraploid cells. Tetraploid and octaploid configurations are established through rejoining of multiple DMA circles.
Figure 4 Models of the DNA structure for diploid (a), tetraploidlzed (b), tetraploid (d), octaploidized (c, e) and octaploid(f) cells. In diploid cells, DMA is connected as homologous chromosomes arranged with point symmetry. Small circles 1 and 6, 2 and 5, and 3 and 4 represent sets of short, medium-sized and long chromosomes, respectively. One to 6 (open circles) and 1* to 6* (closed circles) represent paternal and maternal haploid sets, respectively. When DNA is synthesized without cell division, the DNA twin circles are multiplied, resulting in tetraploidized (b) and octaploidized (c, e) configurations of DNA. Note that the DNA configuration of octaploidized cells is different from the configuration In diploid and tetraploid cells. Tetraploid and octaploid configurations are established through rejoining of multiple DMA circles.Representative cells Some mammalian cells can be polypioidized through DNA synthesis without cell division. Poly-ploidization can be accomplished in a spontaneous fashion or artificially with the use of inhibitors of the cell cycle. Meth-A cells (a methytcholan-threne-induced mouse abdominal dropsy sarcoma cell line) always include a small population of large cells that are produced from diploid cells by spontaneous polyploidization and are eventually removed by apoptosis [17].
Meth-A cells can be polyploidized well by both K-252a and demecolcine, inhibitors of protein kinases and spindle-fiber formation in M phase, respectively [18,19]. Tetraploid Meth-A cell lines have been reproducibly established from diploid Meth-A cells highly polypioidized by demecolcine [20]. Octaploid Meth-A cells were also produced from
tetraploid cells highly polypioidized by demecolcine [21]. Meth-A cells may be susceptible to polyploid transformation, and suitable for studying polypioidized and polyploid celts.
DNA structures of polypioidized and polyploid cells Meth-A cells are highly polypioidized by K-252a; however, most of the celts in a population return to diploid after the drug removal [20]. The tetraploid and octaploid Meth-A cells were established from diploid and tetraploid Meth-A cells, respectively, through polyploidization by demecolcine [20,21]. It seems that there is a distinct difference in genome structure between polypioidized and polyploid cells.
When cell division is inhibited and DNA synthesis progresses, replicated DNA will be stacked (Fig. 4(b), (c) and (e)). When inhibitors are removed, the polypioidized cells may return to the
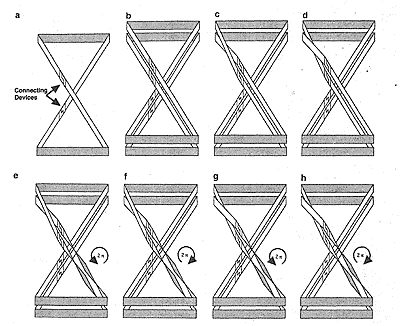 Figure 5 Representative cases for rejoining DNA circles. DNA twin loops are represented as a cross-looped ribbon (a) to facilitate understanding. The looped ribbon contains two couples of devices that can connect ribbons. The DNA ribbon (a) is duplicated and becomes multiple ribbons that cannot be separated without cutting (b). Six cases of joining ribbons are presented (c-h). The resulting configuration of case (g) is the same as that of (a).
Figure 5 Representative cases for rejoining DNA circles. DNA twin loops are represented as a cross-looped ribbon (a) to facilitate understanding. The looped ribbon contains two couples of devices that can connect ribbons. The DNA ribbon (a) is duplicated and becomes multiple ribbons that cannot be separated without cutting (b). Six cases of joining ribbons are presented (c-h). The resulting configuration of case (g) is the same as that of (a). initial ploidy through multiple cell divisions or sequential cell divisions, because the stacked DNA loops have not been linked. When the stacked DNA twin-loops are linked at the center of twin-loop with a proper configuration, the cells may become polyploid cells ((d) and (f) of Fig. 4). Fig. 5 shows the six simple cases of connection between interconnecting DNAs. Only connection G leads to DNA with a configuration similar to that of diploid cells. The cells will be transformed from polyploidized to polyploid cells, suggesting that the polyploidized-polyploid transition involves some difficulty in the connection of interconnecting DNA. The cells in which the connection fails will die.
Devices connecting paternal and maternal DNA in cells We hypothesize the presence of molecular ctevices connecting the paternal and maternal DNA. Unfortunately, there is no experimental evidence for the putative device that joins paternal and maternal DNA. The device may be double-strand DNA which tails an adherent end of single-strand DNA with a specific sequence of basis. Such devices must be
more numerous in polyploid cells, because more
paternal and maternal DNA is connected in polyploid cells.
Ploidy stability in diploid and triploid cells It has been reported that homologous chromosomes are close to each other in germ cells but not in somatic cells [22]. Because homologous chromosomes cannot be in close proximity in somatic diploid cells, homologous chromosomes cannot interfere with the DNA synthesis of the homologous chromosomes, resulting in stable ploidy in diploid somatic cells. On the other hand, if the twin-loops of genome DNA in germ cells are interchanged at junctions by unknowh mechanisms, the homologous chromosomes can be paired in the folded structures. It seems that the DNA configuration is responsible for the stability of ploidy.
The DNA configuration of triploid cells may be a combination of two DNA circles, diploid and haploid DNA circles [23], In triploid cells, the approach of homologous chromosomes is not impossible but is difficult due to the difficulty of achieving a precise fit, and this results in stable ploidy.
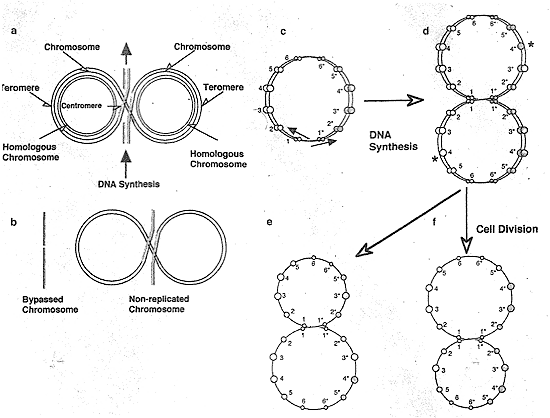 Figure 6 The schema of chromosome loss in tetraploid cells. When DNA replication begins from the midpoint of genome twin circles in tetraploid cells, many of the homologous chromosomes are still paired (c). When DNA replication reaches the paired chromosomes, it may be bypassed by unknown mechanisms (a) and produce bypassed and non-repiicated chromosomes (b). The chromosomes in which mosy such DNA bypasses take place might be conffgurationally decided (d). In this figure, some of the chromosomes belonging to group 4 of the parental and maternal chromosomes are bypassed and missed. The homogeneity of DNA ploidy in the cell population will be maintained (e, f). DNA loss in polyploid cells whose ploidy is a power of 2 Polyploid cells that are produced from diploid cells through DNA replication without cell division have DNA ploidy of a pcwer of two. Let us call such polyploid cells 2n-ploid cells. The homologous chromosomes of 2n-ploid cells are arrayed mirror-symmetrically and they can come close to each other in the folded structure at G1 phase (Fig. (4d) and (f)). If DNA synthesis is bypassed at the paired homotogous chrwnosomes (Fig. 6(a) and (b)), DNA content in 2n-ploid cells is lost at eyery cell division (Fig. 6(d)). As the DNA loss progresses, the chromosome conrfiguration of 2n-ploid cells deviates gradually from mirror-symmetry (Fig. 6(e) and (f)) and the DNA loss ceases, resulting in the establishment semi-stable hypoploid cells such as near-triple cells. This prediction may be supported by the experimental results that near triploid was the terminal ploidy of tetraploid cells [1].
According to the hypothesis that cells adopt specific configuration of DNA, chromosomes in which such DNA bypass mostly takes place might have been configurationally decided. If most cells in cell population undergo such a DNA synthesis bypass at the same homologous chromosomes, a population of celld having equally reduced DNA will be generated/ It will lead to no change in the coefficient of variation (CV) values for the DNA content.
|