В очень сложном головном мозге млекопитающих, также как и в нервных системах более простых организмы, таких как нематода Caenorhabditis elegans, от сотен до тысяч различных классов нейронов может быть выявлено на базе морфологических, физиологических и молекулярных критериев.
Генетический анализ беспозвоночных успешно выявил каскады сигнальных событий, участвующих в дифференцировке специфических типов нейрональных клеток; напр., появление самостоятельных фоторецепторных клеток в глазах Drosophila (rev.Wolff et al., 1997) или спецификация хемосенсорных нейронов у C. elegans (Lanjuin and Sengupta, 2002, Sagasti et al., 1999, Sarafi-Reinach et al., 2001, Sarafi-Reinach and Sengupta, 2000, Troemel et al., 1999). В свою очередь, поиск у позвоночных гомологов Drosophila и C. elegans транскрипционных регуляторов пролил свет на молекулярный контроль нейрогенеза у высших организмов (rev. Livesey and Cepko, 2001, Tanabe and Jessell, 1996). Однако, значительно большая гетерогенность нейронов обнаружена в головном мозге позвоночных, одновременно с отсутствием прямых экспериментальных подходов к идентификации специфических транскриптов, экспрессируемых редкими субпопуляциями нейронов, значительно ограничивают надежды на эти поиски (rev.Cao and Dulac, 2001).
В обонятельном эпителии грызунов каждый из зрелых olfactory sensory
neurons (OSNs) экспрессирует один ген обонятельного рецептора из огромного семейства из почти тысячи генов. Обонятельный эпителий выглядит рыхло организованным в крупные домены экспрессии рецепторов, так что все нейроны, экспрессирующие один и тот же рецепторный транскрипт, по-видимому, случайно диспергированы внутри одной из четырех широких зон, сегрегирующих вдоль дорсо-вентральной оси (Buck and Axel, 1991, Chess, 1994, Malnic et al., 1999, Ressler et al., 1993, Vassar, 1993). Возникают вопросы о регуляции обонятельного предопределения и формирования паттерна эпителия на зоны экспрессии рецепторов.
Чтобы получить непосредственный доступ к сигнальным событиям, связанным с olfactory нейрогенезом, мы разработали технику для характеристики транскрипционных профилей индивидуальных обонятельных нейронов и клеток предшественников (Tietjen et al., 2003). Здесь мы описываем сходную одноклеточную микро-чиповую стратегию, нацеленную на анализ молекулярной спецификации дорсо-вентральных зон экспрессии обонятельных рецепторов. Этот поиск привел к идентификации некоторых транскриптов с дифференциальной экспрессией в разных OR зонах, включая новое семейство генов, названное Lozenge-like (Lzl), которое обнаруживает сходство последовательностей с
Drosophila Lozenge, транскрипционным регулятором, участвующим в формировании паттерна обонятельной и зрительной системы у мух.
Results
General strategy
Olfactory neurogenesis was analyzed at the single-cell level by
comparing the transcriptional profiles of mature olfactory sensory
neurons (OSNs) and immature olfactory neurons (IONs). IONs
are undergoing their differentiation process and display immature
neuronal and olfactory characteristics. Because the respective
ratios of these cell-types change as the animal develops, OSNs
were obtained from adult olfactory epithelium (Tietjen et al.,
2003), while IONs were obtained from olfactory epithelium of
newborn pups. Individual cells were picked at random from
dissociated mouse olfactory epithelia at the respective ages and
single-cell cDNA was obtained as previously described (Dulac
and Axel, 1995). Southern blot analysis was then performed to
ensure that single-cell cDNA samples contained strong expression
of ubiquitous transcripts such as alpha-tubulin and GAPDH, in
addition to verifying the represented cell-type. OSNs were
characterized by strong expression of olfactory marker protein
(OMP) and olfactory receptor (OR), while IONs were identified by
the additional expression of developmental markers such as
PCNA and GAP43 (data not shown). Neither OSNs nor IONs were
found to express the transcription factors Mash1, Ngn1, or NeuroD,
which are markers of earlier olfactory progenitor and precursor
cells. Furthermore, in situ hybridization with the specific olfactory
receptor probe isolated by degenerate PCR from each OSN- and
ION-cDNA enabled us to unambiguously identify the epithelial
zone from which each‘neuron originated (Figure 1A). In total,
cDNAs from 9/45 adult and 10/19 neonatal olfactory epithelium
cells were kept for further analysis and hybridized to Affymetrix
Mu11K high-density oligonucleotide arrays.
Transcriptional profiles of single neurons and sensory
precursors
During the course of our study, amplified single-cell cDNAs
were prepared from a vast collection of sensory neurons including
OSNs, sensory neurons of the adult VNO (VSNs) and
photoreceptors of the retina. In addition, olfactory progenitor cells
(OPCs), which give rise to OSNs, were obtained from E15
olfactory epithelium (Tietjen et al., 2003) and vomeronasal
precursor cells (VPCs) were isolated from E16 VNO. OPCs were
identifed via Southern blot by their strong expression of Mash1,
Ki67 and Cdc2 and by lack of OMP or olfactory receptor expression.
VSNs were identified by the strong expression of OMP, while
VPCs were identified by the expression of Mash1 or Ngn1.
A broad comparison of gene expression across all single cell
cDNAs (Figure 1B) confirms the tight clustering of profiles of cells
of a given type, for example of all mature VNO neurons or of all
olfactory progenitors, while distinct sensory types such as
photoreceptor show considerable divergence. The hierarchical
clustering of the cell profiles (Figure 1B) indicates that VNO and
olfactory neurons, as well as VNO and olfactory precursors, share
more resemblance to each other than each neuron type to its
respective precursor. This clearly emphasizes the similarity
between mature chemosensory neurons together with the
tremendous changes in gene expression occurring during neuronal
development. In contrast, the absence of co-clustering of the
single cell profiles generated from neurons belonging to the same
zone (OSN3, OSN8 and ION6 in zone 4, ION1, ION4 and ION5 in
zone 3, OSN1, OSN6, ION2 and ION3 in zone 2 and OSN5, OSN7
and OSN9 in zone 1) (Figure 1B) suggests that, at least in the data
collected from the 11K murine array, zone-specific transcriptional
differences are likely to be relatively minor. However, despite the
large number of developmental differences among different celltypes,
the extreme sensitivity of single-cell transcriptional profiling
still allowed us to identify several zone-specific transcriptional
differences, as shown below.
Specific transcription in olfactory zones and the lozenge-like
family
We reasoned that subtle changes in gene expression in mature
olfactory neurons from different MOE zones could be identified by
comparing the transcriptional profiles of single OSNs.
Transcriptional profiles of OSNs originating from adult MOE
(OSNs 1-9) have been described previously (Tietjen et al., 2003).
Using the same single-cell microarray method, we also obtained
the transcriptional profiles of several OSNs from neonatal MOE
(ION 1-7). The quality of cDNA samples representing OSNs and
IONs was determined based on the strong expression of alphatubulin
and GAPDH by Southern blot and the detection of a single
olfactory receptor sequence by degenerate PCR. Following
microarray hybridization, however, the average pairwise correlation
coefficients of gene expression were considerably lower for
neonatal IONs than for adult OSNs (0.53 +/- 0.01 versus 0.68 +/
- 0.05), indicating a greater level of transcript heterogeneity
among neonatal cells. Indeed, Southern blot and microarray
analyses indicate that neonatal IONs have variable expression of
markers of cell division (e.g., PCNA), axon growth (e.g., GAP43)
and other developmentally-regulated events (e.g., RGS11, Sox11;
data not shown), suggesting that these cells are still undergoing
various steps of neuronal maturation.
To determine spatial differences in MOE neuronal gene expression,
we first identified the original zone of each olfactory neuron using its
olfactory receptor sequence as a probe for RNA in situ hybridization.
Each olfactory receptor sequence gave robust expression in an
apparently random subset of OSNs confined to one zone (data not
shown). Based on 3-4 cells attributed to each zone, we then
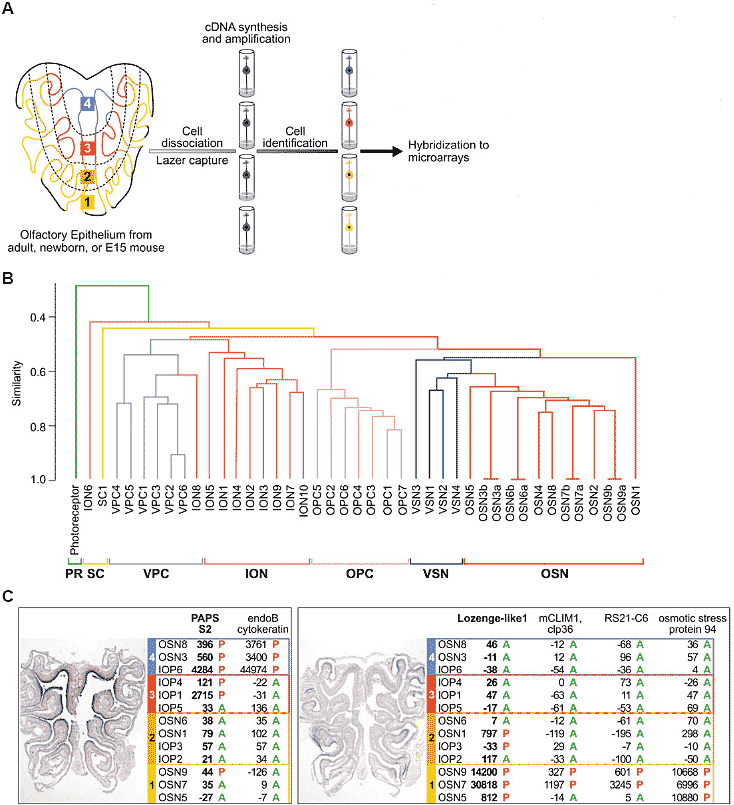 Fig. 1. Monitoring transcription in single olfactory neurons. (A) Strategy. The olfactory epithelium of rodent is subdivided into four broad zones
(colored numbers and lines), each characterized by the expression of a subset of olfactory receptor genes. In addition, each epithelium comprises cells at various stages of neuronal development. Olfactory epithelia dissected from adult, newborn, or e15 mice were dissociated. Individual olfactory neurons and olfactory precursors were picked at random and seeded into individual PCR tubes before undergoing cell lysis, cDNA synthesis and PCR amplification. The zone of origin and the developmental stage of each cell were retrospectively determined by PCR amplification of olfactory receptor genes present in each single cell cDNA and by Southern blot hybridization with developmental- and olfactory-specific markers. cDNA samples representing neurons or precursors of interest were then hybridized to Affymetrix Mu11K GeneChips. (B) Dendrogram showing the hierarchical clustering of single-cell samples based on probe set intensity. Samples of the same cell-type tend to have similar transcriptional profiles: Mash1-positive OSNs, IONs, OPCs, vomeronasal sensory neurons (VSNs) and vomeronasal precursor cells (VPCs) tend to be more related to each other than to other cell types, with the exception of some IONs (see ION6 and ION8). All olfactory cell types are considerably different from photoreceptors. Similarity was calculated based on the Pearson’s correlation coefficients among samples. Abbreviations: a,b, same cell sample hybridized to duplicate microarrays.
SC, supporting cell. (C) Microarray prediction of the expression patterns of novel transcripts based on single cell profiles. Transcripts with expression patterns in a subset of olfactory receptor zones were identified based on differential expression in single OSNs and IOPs originating from specific zones. For each transcript, the ADV and Presence/Absence call in single cells from each zone are shown. For
example, the specific expression of a novel 3’-splice variant of PAPS-S2/SK2 in the dorsal olfactory epithelium was predicted from the differential expression in neurons originating from zones 3 and 4, as shown by RNA in situ hybridization (left). Similarly, Osp94, mCLIM1/Clp36 and two transcripts of unknown function were expressed preferentially in zones 1 and 2 (right).
Fig. 1. Monitoring transcription in single olfactory neurons. (A) Strategy. The olfactory epithelium of rodent is subdivided into four broad zones
(colored numbers and lines), each characterized by the expression of a subset of olfactory receptor genes. In addition, each epithelium comprises cells at various stages of neuronal development. Olfactory epithelia dissected from adult, newborn, or e15 mice were dissociated. Individual olfactory neurons and olfactory precursors were picked at random and seeded into individual PCR tubes before undergoing cell lysis, cDNA synthesis and PCR amplification. The zone of origin and the developmental stage of each cell were retrospectively determined by PCR amplification of olfactory receptor genes present in each single cell cDNA and by Southern blot hybridization with developmental- and olfactory-specific markers. cDNA samples representing neurons or precursors of interest were then hybridized to Affymetrix Mu11K GeneChips. (B) Dendrogram showing the hierarchical clustering of single-cell samples based on probe set intensity. Samples of the same cell-type tend to have similar transcriptional profiles: Mash1-positive OSNs, IONs, OPCs, vomeronasal sensory neurons (VSNs) and vomeronasal precursor cells (VPCs) tend to be more related to each other than to other cell types, with the exception of some IONs (see ION6 and ION8). All olfactory cell types are considerably different from photoreceptors. Similarity was calculated based on the Pearson’s correlation coefficients among samples. Abbreviations: a,b, same cell sample hybridized to duplicate microarrays.
SC, supporting cell. (C) Microarray prediction of the expression patterns of novel transcripts based on single cell profiles. Transcripts with expression patterns in a subset of olfactory receptor zones were identified based on differential expression in single OSNs and IOPs originating from specific zones. For each transcript, the ADV and Presence/Absence call in single cells from each zone are shown. For
example, the specific expression of a novel 3’-splice variant of PAPS-S2/SK2 in the dorsal olfactory epithelium was predicted from the differential expression in neurons originating from zones 3 and 4, as shown by RNA in situ hybridization (left). Similarly, Osp94, mCLIM1/Clp36 and two transcripts of unknown function were expressed preferentially in zones 1 and 2 (right).
searched the microarray data for transcripts predicted to be enriched
in the ventral-most (Zone 1) or dorsal-most (Zone 4) zone of the MOE
(Figure 1A). As zone-specific transcripts are likely to represent a very
low percentage of the total transcripts expressed by neonatal and
adult OSNs, while developmental and other cell-intrinsic differences
of OSNs are likely to represent a much higher percentage (Figure
1B), we observed that a large portion of candidate zone-specific
transcripts represented false-positive signals. However, using a
standard hierarchical clustering approach (Eisen et al., 1998; see
Experimental Procedures), two factors were successfully identified
as preferentially expressed in the most dorsal zones of the adult
MOE, while four transcripts were identified in the most ventral zones
(Figure 1C). Similar results were also obtained using self-organizing
map and confidence interval approaches (Tamayo et al., 1999;
Tietjen et al., 2003; data not shown). Predictions made from the
arrays were confirmed by in situ hybridization. Two transcripts were
identified that display preferential expression in the most dorsal
zones of the olfactory epithelium. One encodes a novel 3’ splice
variant of the sulfate-activating enzyme PAPS synthase2/SK2 and is
expressed in a gradient with zone 4 showing the highest expression
level (Figure 1C). The function of this gene in the adult olfactory
epithelium is unknown but one may suspect that this enzyme activity
could be related with differential glucoconjugate synthesis (Kurima et
al., 1998).
Conversely, several transcripts, including one encoding an alphainterferon
induced protein of unknown function, were found to be
strongly and differentially expressed in the ventral zones of the
olfactory epithelium (Figure 1C). This latter transcript captured our
attention because it encodes a protein which displays distant but
noticeable similarity with the runt domain-containing protein Lozenge,
a transcription factor involved in the prepatterning of the Drosophila
eye and olfactory organs and in the regulation of the proneural gene
amos in olfactory sensillae (Canon and Banerjee, 2000, Flores et al.,
1998, Goulding et al., 2000). A 29% sequence similarity to Lozenge
was found over a stretch of 147 amino acids encompassing part of
the conserved DNA-binding and transactivating portions of the
Lozenge runt domain (data not shown). Database searches and
screening of a mouse olfactory cDNA library revealed a total of three
mouse genes predicted to encode proteins that share a common
motif similar to part of and extending downstream of the Lozenge runt
domain. These genes are thus named Lozengelike 1-3 (Lzl1-3,
Figure 2A). Similar searches also revealed numerous splice variants
of Lzl1 and Lzl2 in mouse, including some that lack the conserved Lzl
domain (Figure 2B). While most Lzl1 and Lzl2 sequences are
predicted to contain one Lzl motif, Lzl3 is predicted to contain two
motifs (Figure 2A). Lzl1-3 were all identified within a ~23 kb genomic
region of mouse Chromosome 12E (data not shown), suggesting that
they arose from recent gene duplication events. Although divergent
Lzl sequences were identified in human, rat and zebrafish databases
(data not shown), database searches revealed no obvious similarities
to other genes or other known protein motifs.
Strikingly, two members of the Lzl family display zone-restricted
expression in adult MOE. Lzl1a, the transcript originally identified by
microarray, is specifically expressed in Zones 1 and 2, as determined
by expression of olfactory receptor sequences with established zone
identities on adjacent sections (Figure 2C). All Lzl1 splice variants
investigated gave identical expression patterns (n=5, data not shown).
In contrast, Lzl2a is detected in a subset of basal cells and underlying
lamina propria found exclusively in the dorsal MOE (Figure 2C). By
in situ hybridization, we estimate the onset of detectable expression
to the first postnatal week. Multiple splicing variants of lozenge-like1
were identified in the olfactory cDNA library while the expression of
lozenge-like3 was not detected in this tissue. In addition, lozengelike1
and 2 were found by Northern blot analysis in a variety of tissues
including adult lung, liver, spleen and heart, indicating that their
expression is not olfactory specific (data not shown).
Discussion
Нейроны представляют собой лишь минимальный компонент сенсорных органов и внутри каждого сенсорного эпителия небольшая субпопуляция рецепторных клеток специализируется морфологически и молекулярно для обнаружения ограниченного круга средовых сигналов. Субнаборы фоторецепторных клеток в сетчатке специализируются в детекции фотонов определенных длин волн, а волосковые клетки внутреннего уха образуют специфические конфигурации стереоцилий и ионные токи обеспечивают тонкую настройку на частоты звуков. Сходным образом, обонятельный эпителий млекопитающих содержит высоко гетерогенную и постоянно обновляющуюся популяцию нейронов и нейрональных предшественников. У мышей каждый зрелый olfactory sensory neuron (OSN) экспрессирует уникальный обонятельный рецепторный ген из семейства почти в тысячу генов, а все нейроны, экспрессирующие один и тот же рецепторный транскрипт случайно разбросаны внутри одной из 4-х широких зон обонятельного эпителия (Buck and Axel, 1991, Chess, 1994, Malnic et al., 1999, Ressler et al., 1993, Vassar, 1993).
Какие транскрипционные регуляторы лежат в основе такого сенсорного разнообразия? Генетический анализ сетчатки у мышей, внутреннего уха и обонятельного эпителия подчеркивает важную роль транскрипционных факторов, содержащих basic helix-loop-helix (bHLH), родственных белкам Drosophila achaete-scute и atonal, в контроле широкого сенсорного спектра нейрональных предшественников (rev. Bertrand et al., 2002). Предшественники внутреннего уха экспрессируют atonal гомолог Math1, а мыши с делецией Math1 не способны формировать зрелые внутренние волосковые клетки (Bermingham et al., 1999). Сходным образом, зрелые обонятельные сенсорные нейроны не образуются у мышей с делецией achaete-scute гомолога Mash1 (Guillemot et al., 1993). Экспрессия Mash1 в ранних olfactory progenitor cells (OPCs) контролирует экспрессию bHLH содержащих транскрипционных факторов Ngn1 и NeuroD, которые в свою очередь регулируют обонятельную дифференцировку (Cau et al., 1997). Однако, как внутри данной modality, происходит приобретение особенных транскрипционных регуляторов сенсорными предшественниками, что создает сенсорное разнообразие, например, чтобы выбрать специфический обонятельный рецептор, тип opsin или изоформу ионного канала, остаётся в основном неясным в первую очередь из-за того, что клетки предшественники представлены так редко и из-за разнородности популяции в развивающемся сенсорном эпителии. Спустя более 10 лет после открытия огромного семейства, кодирующего обонятельные рецепторы (Buck and Axel, 1991), механизм, с помощью которого одиночный обонятельный нейрон выбирает экспрессию уникального рецепторного гена, по-прежнему непонятен. Сформулированы различные гипотезы с вовлечением экспрессии сложных комбинаций транскрипционных факторов или с существованием соматической рекомбинации или ремоделированием хроматина для позиционирования регуляторного элемента выше избранного рецепторного гена. Независимо от точного механизма выбора одиночного рецептора простое существование дорсо-вентрального паттерна экспрессии рецепторов в 4-х зонах обонятельного эпителия указывает на механизм пространственной передачи сигналов внутри эпителия.
Как могут быть идентифицированы регуляторы зон экспрессии рецепторов? В первом приближении мы решили исследовать различия в экспрессии генов
 Fig. 2. The Lozengelike family. (A) Amino acid sequence alignment of Lzl1-3. The conserved region with similarity to the Drosophila Lozenge transcription factor is underlined in red. Lzl3 also has a second conserved region, underlined in blue. (B) Schematic of the alignment of Lzl1 and Lzl2 splice variants. Overlapping amino acid sequences are shown in grey. The conserved Lzl domain is shown in blue. The putative start Methionine, when known, is indicated (M). (C) Expression patterns of Lozengelike1a and 2a in the dorsal (zone 4) and ventral (zone 1) portions of adult MOE. Lozengelike 1a is expressed throughout the ventral MOE, while Lozengelike 2a is found only in basal cells and underlying lamina propria of the dorsal MOE.
Fig. 2. The Lozengelike family. (A) Amino acid sequence alignment of Lzl1-3. The conserved region with similarity to the Drosophila Lozenge transcription factor is underlined in red. Lzl3 also has a second conserved region, underlined in blue. (B) Schematic of the alignment of Lzl1 and Lzl2 splice variants. Overlapping amino acid sequences are shown in grey. The conserved Lzl domain is shown in blue. The putative start Methionine, when known, is indicated (M). (C) Expression patterns of Lozengelike1a and 2a in the dorsal (zone 4) and ventral (zone 1) portions of adult MOE. Lozengelike 1a is expressed throughout the ventral MOE, while Lozengelike 2a is found only in basal cells and underlying lamina propria of the dorsal MOE.
между нейронами, принадлежащими к разным зонам во взрослом эпителии. Зрелый обонятельный эпителий является особенно впечатляющим примером составной нейрональной структуры, в которой предшественники на разных стадиях онтогенеза, а также тысячи нейрональных типов перемешаны и группируются в 4 широкие зоны эпителия. Чрезвычайно закрученные (convoluted) структуры обонятельных носовых конх (turbinates) у грызунов мешают любой попытке исследовать молекулярные различия между OR зонами с помощью хирургического иссечения эпителия. Однако, идентификация обонятельных рецепторов из каждой OSN кДНК позволила нам в свою очередь определить происхождение из OR зон для каждого нейрона и тем самым установить транскрипционные различия между обонятельными нейронами, принадлежащими к разным OR зонам. Несмотря на предсказания, что различия между зонами будут представлять минорная часть клеточных профилей, но чрезвычайная чувствительность и разрешение на клеточном уровне представили профили одиночных клеток как способные в действительности идентифицировать гены с дифференциальной экспрессией между нейронами, принадлежащими к разным OR зонам. Наш поиск идентифицировал несколько очень родственных генов, названных здесь lozenge-like 1 - 3, которые облают общими мотивами внутри домена lozenge белка, который представляет собой части из и распространяется 3' на runt домен (Canon and Banerjee,
2000, Flores et al., 1998, Goulding et al., 2000). Функция этих генов неизвестна, но достоверные различия внутри канонических runt доменовых мотивов делают маловероятным, что lozenge-like белки играют роль истинных runt домен-содержащих транскрипционных факторов. Поразительно, два члена этого семейства обладают ограниченной экспрессией в зонах обонятельного эпителия:
lozenge-like 1 специфически экспрессируется в вентральном эпителии, тогда как
lozenge-like 2 обнаруживается в субнаборе базальных клеток, обнаруживаемых исключительно в дорсальном обонятельном эпителии. Необходимы дальнейшие исследования для описания функции семейства Lzl и для определения, обладают ли они какими-либо функциональными свойствами формирования сенсорного паттерна вместе со своим ближайшим гомологом Lozenge в геноме дрозофилы.
Широко-массштабный анализ транскрипционных профилей, полученных от индивидуальных обонятельных нейронов и обонятельных предшественников предоставляет уникальный моментальный снимок различных регуляторных сетей, конкурирующих или борющихся внтри одиночной клетки на специфической стадии развития за контроль клеточной пролиферации и обонятельной спецификации. В целом, получение профилей одиночных клеток, представленное здесь открывает беспрецендентный уровень чувствительности и клеточного разрешения, который позволит выяснить молекулярные основы нейрональной специфичности в головном мозге.
Сайт создан в системе
uCoz
