Epigenetic systems in mammalian development
У одноклеточных организмов каждая индивидуальная клетка использует почти всю геномную информацию и располагает очень идентичным фенотипом. Напротив. у млекопитающих имеется, по крайней мере, несколько сотен, различных типов клеток. Все эти типы клеток происходят из одной оплодотворенной яйцеклетки. Дифференцировка каждого типа клеток достигается без изменения последовательностей ДНК, на за счет скоординированного использования субнаборов генов. Для этого используется ряд эпигенетических механизмов (Shiota, 2004; Lieb et al., 2006).
У млекопитающих метилирование ДНК осуществляется посредством DNA
methyltransferases (Dnmts), которые оперируют внутри 5'-CG-3' динуклеотидов (CpG: C сопровождаемый G). 5-Methylcytosine (5-MeC) является единственной химической модификацией геномной ДНК млекопитающих. Статус метилированных CpGs сохраняется и после репликации ДНК с помощью Dnmts и , следовательно, наследуется в чреде клеточных поколений (Bird, 2002). 5-MeC обнаруживается в большей части ДНК эукариот, включая грибы, растения и позвоночных (Bird, 2002). Прототип Dnmt обнаружен и в одноклеточных организмах, таких как Escherichia coli. Dnmt гены законсервированы почти у всех эукариот, однако 5-MeC отсутствует или редок у видов грибов (Saccharomyces cerevisiae, Saccharomyces pombe), мух (Drosophila melanogaster) и нематод (Caenorhabditis elegans, Pristionchus pacificus) из-за делеции гена или мутаций, которые тяжело нарушили ферментативную активность (Goll and Bestor, 2005). Следовательно, адаптация и/или поддержание системы метилирования ДНК может быть ключевым молекулярным событием в эволюции в отношении дивергенции видов и происхождения позвоночных.
Гистоновые модификации др. форма эпигенетической регуляции, которая соответствует пост-трансляционным модификациям N-терминальных хвостов гистонов. Так, свыше 60 различных остатков стержневых гистонов (H2A, H2B, H3 и H4) могут быть модифицированы. Модификации включают ацетилирование, метилирование, фосфорилирование, убиквитилирование, сумоилирование, ADP рибозилирование, deimination и изомеризация proline (Kouzaridez, 2007). Некоторые гистоновые модификации служат, чтобы координировать наборы 5-MeC.
Эти эпигенетические маркеры обычно ассоциируют с конденсацией хроматина, которая играет критическую роль в молчании генов, стабилизации хромосомной структуры и супрессии мобильности ретротранспозонов.
Клеточная дифференцировка использует массивные альтерации эпигенетического статуса по всему геному во множественных генных локусах с изменениями как de novo метилирования и деметилирования ДНК, так и различных гистоновые модификации (Ohgane et al., 2002; Shiota et al., 2002; Eckhardt et al., 2006; Bracken et al., 2006; Squazzo et al., 2006; Oakes et al., 2007; Mikkelsen et al., 2007). В самом деле, имеются многочисленные ткане-зависимые, дифференциально метилированные регионы ДНК (T-DMRs) в уникальных последовательностях, включая гены и их регуляторные элементы в геноме млекопитающих.
Tissue-dependent, differentially methylated regions (T-
DMRs) in unique sequences in the mammalian genome
DNA methylation in repetitive and unique sequences
Удивительным свойством генома млекопитающих является то, что огромная его порция представлена не-генными, повторяющимися элементами, включая вкрапленные повторы и сателлиты: ~49% и 44% в геноме человека (hg18 assembly) и мыши (mm9 assembly) соотв. (Repeatmasker; http://www.repeatmasker.org/). Свыше 60% CpGs генома мыши метилированы, большинство из которых локализуется в повторяющихся элементах (Solage and Cedar, 1978; Gruenbaum et al., 1981). Метилирование ДНК повторяющихся элементов может функционировать, чтобы супрессировать транспозоны и усиливать стабильность генома (Yoder et al., 1997).
До 1980s было найдено несколько генов, которые метилируются (McGhee and Ginder, 1979), о они рассматривались как исключительные локусы, поскольку уровень 5-MeC в уникальных последовательностях был намного ниже, чем в повторяющихся последовательностях (Ehrlich et al., 1982). Однако недавние исследования установили, что метилирование ДНК используется для регуляции значительно большего количества генов, экспрессируемых ткане-специфическим и клеточно-специфическим образом и тем самым играющее роль в дифференцировке и развитии (Shiota, 2004; Yagi et al., 2008). Эпигенетическая динамика генов отличается от это массы ДНК. Недавно серьезно внимание было привлечено к эпигенетической регуляции генов. Сегодня известно ~25,000 генов, описанных для генома человека (hg18) и мыши (mm9) , которые представляют менее 3% генома (UCSC genome browser; http://genome.ucsc.edu/). До сих пор были обнаружены различные гены с T-DMRs .
DNA methylation profiles of T-DMRs in normal cells and tissues
Исследования по всему геному показали, что геном млекопитающих содержит многочисленные T-DMRs в его уникальных последовательностях. Разные техники были использованы, чтобы получить профили метилирования ДНК уникальных последовательностей, включая: restriction land mark genomic scanning (RLGS) (Ohgane et al., 2002), genome-wide bisulfite sequencing (Eckhardt el al., 2006) и микромассивы в комбинации с рестрикционными энзимами, чувствительными к метилированию ДНК (Khulan et al., 2005; Yagi et al., 2008) или антителами против 5-MeC (Weber et al., 2005). Все эти исследования сообщили об существенном количестве T-DMRs путем сравнения профилей метилирования ДНК из различных соматических тканей (Ohgane et al., 2002; Shiota et al., 2002; Kremenskoy et al., 2003; Song et al., 2005; Khulan et al., 2006; Eckhardt et al., 2006; Sakamoto et al., 2008; Yagi et al., 2008), стволовых клеток (Shiota et al., 2002; Kremenskoy et al., 2003), зародышевых клеток (Shiota et al., 2002; Oakes et al., 2007; Weber et al., 2007), первичных типов клеток (Weber et al., 2005; Eckhardt et al., 2006; Weber et al., 2007), и клеток от разных полов и возрастов (Weber et al., 2005; Eckhardt et al., 2006). Напр., RLGS с Not I, чувствительным к метилированию рестрикционным энзимом, был использован для анализа статуса метилирования ДНК по всему геному (~1,500 loci) embryonic stem (ES) клетках мышей, embryonic germ (EG) клетках,
trophoblast stem (TS) клетках, зародышевых клетках и некоторых соматических тканях, при этом идентифицировано 247 T-DMRs, в которых статус метилирования зависел от типа клеток или ткани (Shiota et al., 2002). Итак, каждый тип клеток/ткани обладает специфическим профилем метилирования T-DMRs, и эти профили могут быть использованы для различения типов клеток/тканей (Fig. 1).
Исходя из того факта, что приблизительно 70% Not I сайтов были локализованы внутри CpG островков в геноме мыши (Fazzari and Greally, 2004), было предположено, что CpG островки содержат T-DMRs. Однако такой тип T-DMRs представляет собой относительно небольшую пропорцию (Eckhardt et al., 2006; Sakamoto et al., 2007). Кроме того, T-DMRs в CpG островках в целом ограничены субрегионами. Напр., T-DMR из гена Sphk1, который законсервирован у мышей, крыс и человека, занимает ~200 base pairs (bp) на краю островка CpG в 3.7-kilo bp, фракция менее 10% (Imamura et al., 2001). Такой тип T-DMR, который ограничен регионом небольших CpG островков, также был найден в EDNRB и POMC генах (Shiota, 2004). С др. стороны, все CpGs в CpG островке метилированы в Tact1/Actl7b и
Ant4/Slc25a31 генах в соматических тканях, тогда как они неметилированы в зародышевых клетках (Hisano et al., 2003; Suzuki et al., 2007). Следовательно, имеется новый тип CpG островковых T-DMR, при которых все CpGs могут быть полностью метилированы в нормальных клетках.
Недавно анализ по всему геному подтвердил, что существуют регионы с низким содержанием CpG также как и те, что имеют высокие частоты CpG, включая CpG островки, которые фактически метилированы (Eckhardt et al., 2006; Weber et al., 2007; Sakamoto et al., 2007; Fouse et al., 2008). Итак, существуют T-DMRs, ассоциированные с генами как бедными CpG, так и богатыми CpG последовательностям. В ближайшее время количество T-DMRs д. увеличиться с расширением исследований и профиль метилирования ДНК в целом геноме окажется более сложным.
Профиль метилирования ДНК специфичен для типов клеток и тканей,
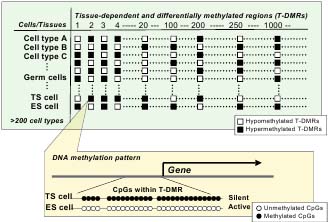 Fig. 1. Cell-type-specific DNA methylation profiles consisting of differentially methylated T-DMRs. Each cell type (ES cells and TS cells) has a unique pattern of DNA methylation at CpGs within T-DMRs (lower panel). These differential methylation status in T-DMRs constitute cell-type-specific DNA methylation profiles genome-wide (upper panel). The DNA methylation profile of a cell type can be used as a novel tool to define and characterize the cell type.
Fig. 1. Cell-type-specific DNA methylation profiles consisting of differentially methylated T-DMRs. Each cell type (ES cells and TS cells) has a unique pattern of DNA methylation at CpGs within T-DMRs (lower panel). These differential methylation status in T-DMRs constitute cell-type-specific DNA methylation profiles genome-wide (upper panel). The DNA methylation profile of a cell type can be used as a novel tool to define and characterize the cell type.
подобно отличиям отпечатков пальцев, и может быть использован как identification (ID) метка для клеток (Fig. 1). Профиль метилирования ДНК также предоставляет новый метод оценки сходства клеток, поскольку он отражает сходство в паттерне дифференцировки клонов (Sakamoto et al., 2007). Поскольку профиль метилирования ДНК является уникальным ID клеток, то изменения профиля метилирования ДНК д. вызывать альтерации клеточных признаков.
Epigenetic system of DNA methylation and histone
modification
DNA methylation, histone modification and chromatin structure
Статус метилирования ДНК тесно связан со структурой хроматина (Fig. 2). Известно, что фракция к micrococcal nuclease-резистентного закрытого хроматина преимущественно содержит 5-MeC (Solage and Cedar, 1978). Гистоновые модификации также влияют на структуру хроматина. Ацетилирование остатков лизина, как полагают, непосредственно нейтрализует позитивный заряд гистонов и вносит вклад в реляксацию взаимодействия между ДНК и гистонами.
Хотя это не всегда имеет место, но исследования in vitro показали, что ацетилирование в K16 гистона H4 достаточно, чтобы ингибировать образование 30 nanometer-like хроматиновых волокон (Shogren-Knaak et al., 2006). Др. путь обеспечивает распознавание белками гистоновых модификацией, включая chromodomain и bromodomain
белки, которые соединяются с метилированными и ацетилированными лизинами, соотв., и влияют на структуру хроматина как члены комплексов транскрипционных репрессоров или ремоделирующих хроматин (Kouzaridez, 2007). Ацетилирование гистонов H3/H4 (H3Ac, H4Ac)
и ди- и триметилирование лизина 4 из H3 (H3K4me2 и me3) обычно обнаруживается в транскрипционно активном, истощенному по нуклеосомам и чувствительном к нуклеазе хроматине (Birney et al., 2007). Напротив метилирование K9 и K27 гистона
H3 (H3K9me и K27me) обычно обнаруживается в транскрипционно молчащих регионах хроматина (Lachner et al., 2003). Профиль гистоновых модификаций в неповторяющихся регионах генома также уникален для типа клеток или ткани (Hattori et al., 2004b; Bracken et al.,
2006; Squazzo et al., 2006; Tomikawa et al., 2006; Mikkelsen et al., 2007).
Epigenetic control of stemness-related genes (Oct-4 and Nanog)
В эмбриогенезе млекопитающих первое событие дифференцировки, которое детерминирует клон трофэктодермы и inner cell mass (ICM) происходит на ст. бластоциста. ES клетки, произошедшие от ICM обнаруживают плюрипотентную способность вносить вклад во все эмбриональные клоны (Martin, 1981). TS клетки, произошедшие от трофэктодермы обладают способностью дифференцироваться в клон трофобласта in vitro (Tanaka et al., 1998). Ген Oct-4 является членом семейства POU транскрипционных факторов и обладает CG-богатым и TATA-less промотором. Oct-4 экспрессируется в ES клетках, но не в TS клетках (Tanaka et al., 1998) а снижение экспрессии гена Oct-4 вызывает трансдифференцировку ES клеток в TS-подобные клетки при определенных условиях культивирования (Niwa et al., 2000). Имеется T-DMR в промоторном и энхансерном регионе гена Oct-4 и T-DMR гипометилирован в ES клетках, но гиперметилирован в TS клетках (Hattori et al., 2004b). ДНК гипометилирование T-DMR в гене Oct-4 ассоциирует с гиперацетилированием гистонов в ES клетках, указывая тем самым, что статус метилирования ДНК коррелирует со структурой хроматина (Hattori et al., 2004b). Nanog является др.транскрипционным фактором, который связан со стволовостью (stemness) преимплантационных эмбрионов и ES клеток.
Ген Nanogтакже имеет T-DMR с относительно богатой CpG последовательностью (Hattori et al., 2007). Подобно Oct-4, Nanog T-DMR гиперметилирован в TS клетках и гипометилирован в ES клетках. Nanog T-DMR также гиперацетилирован в ES клетках и гипоацетилирован в TS клетках. В Nanog T-DMR из TS клеток lysines 9 и 27 (K9 and K27) гистона H3 гиперметилирован. Напротив, метилирование и K9 и K27 низкое в Oct-4 T-DMR. Т.о., эти гены регулируются с помощью интимных взаимоотношений между метилирование ДНК и модификациями гистонов в разных комбинациях.
Epigenetic factors involved in epigenome formation
Геном млекопитающих обладает сложным профилем метилирования ДНК и гистоновых модификаций, это обозначается как эпигеном. Профили четко показывают, что дифференцировка использует массивные альтерации эпигенетического статуса по всему геному во множественных генетических локусах, с изменениями как в метилировании ДНК, так и в различных гистоновых модификациях (Ohgane et al., 2002; Shiota et al., 2002; Eckhardt et al., 2006; Bracken et al., 2006; Squazzo et al., 2006; Oakes et al., 2007; Mikkelsen et al., 2007). Возникает вопрос, как эти профили устанавливаются в соотв. типе клеток и в соотв. время и как эта информация наследуется в последующих клеточных поколениях. Принимая во внимание взаимодействия между метилированием ДНК и гистоновыми модификациями, напрашивается один механизм, лежащий в основе эпигенетической памяти (Birke et al., 2002; Fujita et al., 2003; Fuks et al., 2003a; Esteve et al., 2006; Li et al., 2006a; Vire et al., 2006; Ikegami et al., 2007). Эти эпигенетические факторы и их взаимодействие суммированы в этом разделе (Fig. 2).
DNA methyltransferases
У млекопитающих существуют три функциональные DNA methyltransferases (Dnmts), Dnmt1, Dnmt3a и Dnmt3b.
Dnmt1 и Dnmt3bнокаутные мыши являются эмбриональными леталями на ст. (E) 8.5 и 14.5-18.5, соотв., тогда как нокауты
Dnmt3a
погибают в возрасте 4 недель (Li et al., 1992; Okano et al., 1999), указывая тем самым на важную роль метилирования ДНК для развития млекопитающих . Dnmts были подразделены на две группы, исходя из их предпочтения к субстратам. Dnmt1 преимущественно добавляет метильную группу к полу-метилированной ДНК (метилирована только одна из двойных нитей) по сравнению с неметилированной формой
in vitro (Gruenbaum et al., 1982). Это указывает на то, что Dnmt1 метилирует дочернюю нить во время репликации, чтобы сохранить родительский паттерн метилирования ДНК. Фактически, Dnmt1 локализуется в фокусах репликации во время S фазы (Leonhardt et al., 1992) и непосредственно взаимодействует с methyl-CpG-binding protein 2 (MeCP2), который распознает полу-метилированную ДНК (Kimura and Shiota, 2003). Поэтому Dnmt1 была названа "maintenance methyltransferase" благодаря её вкладу в наследование меток метилирования ДНК в поколениях. Др. класс содержит Dnmt3a и Dnmt3b, которые предпочитают неметилированную ДНК в качестве субстрата in vitro (Okano et al., 1998). Кроме того, двойной нокаут Dnmt3a и Dnmt3b предупреждает
de novo метилирование ДНК, индуцированное с помощью вирусной инфекции. Поэтому Dnmt3a и Dnmt3b были названы "de novo methyltransferases" (Okano et al., 1999). Однако такая классическая группировка по поддержанию и de novo метилированию может быть неадекватной для понимания внутриклеточных механизмов, поскольку существуют прямое взаимодействие между DNMT1, 3A aи 3B
in vivo, как это было показано на клетках человека (Kim et al., 2002). Нокаут Dnmt3a и Dnmt3b показывает прогрессирующую потерю метилирования ДНК в повторяющихся последовательностях с увеличением количества пассажей, указывая, что Dnmt3a и Dnmt3b также функционируют и как "maintenance methyltransferases" (Chen et al., 2003). Ряд обычно метилированных уникальных последовательностей вызывает резкую потерю метилирования ДНК в Dnmt3a и Dnmt3b двойных нокаутных ES клетках, тогда как только частичное снижение достигается в Dnmt1 нокаутных клетках (Hattori et al., 2004a). Напротив, уровни метилирования ДНК в повторяющихся последовательностях
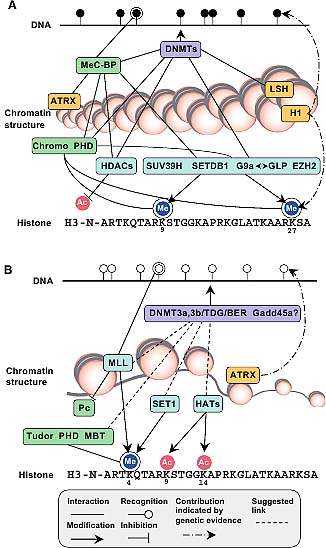 Fig. 2. Molecular link between DNA methylation, chromatin structure and histone modifications in inactive (A) and active (B) genomic regions. BER, base excision repair proteins (e.g. AP endonuclease, DNA polymerase I); Chromo, chromodomain proteins (e.g. HP1, Pc); DNMTs, DNA methyltransferases (e.g. Dnmt1, Dnmt3s); HATs, histone acetylases (e.g. CBP/P300); HDACs, histone deacetylases (e.g. HDAC1, HDAC2); MBT, MBT domain proteins; MeC-BPs, methylCpG binding proteins (e.g. MBDs, MeCP2, UHRF1); PHD, PHD finger proteins (e.g. BPTF, SMCX); TDG, thymine DNA glycosylase; Tudor, Tudor domain
proteins.
Fig. 2. Molecular link between DNA methylation, chromatin structure and histone modifications in inactive (A) and active (B) genomic regions. BER, base excision repair proteins (e.g. AP endonuclease, DNA polymerase I); Chromo, chromodomain proteins (e.g. HP1, Pc); DNMTs, DNA methyltransferases (e.g. Dnmt1, Dnmt3s); HATs, histone acetylases (e.g. CBP/P300); HDACs, histone deacetylases (e.g. HDAC1, HDAC2); MBT, MBT domain proteins; MeC-BPs, methylCpG binding proteins (e.g. MBDs, MeCP2, UHRF1); PHD, PHD finger proteins (e.g. BPTF, SMCX); TDG, thymine DNA glycosylase; Tudor, Tudor domain
proteins.
сильно нарушены при Dnmt1 недостаточности (Li et al., 1992). Следовательно, Dnmts обладают предпочтением к определенным характеристикам последовательностей мишеней скорее, чем к последовательному процессу установления и поддержания метилирования ДНК с помощью Dnmt3a/3b , сопровождаемому Dnmt1. Недавнее исследование показало, что Dnmt3a/3b способны deaminate 5-MeC в двунитчатой ДНК, это генерирует T:G совпадения (mismatch) (Metivier et al., 2008). Скоординированное рекрутирование Dnmt3a/3b,
thymine DNA glycosylase (TDG) и base excision repair (BER) белков происходит во время деметилирования ДНК гена мишени estrogen receptor alpha (ERα) , pS2 промотора, который обладает периодическим метилированием/деметилированием ДНК во время циклов транскрипции, индуцируемых с помощью estrogen (Metivier et al., 2008; Kangaspeska et al., 2008). Эти результаты указывают на то, что Dnmt3a/3b, в кооперации с TDG и BER белками играют активную роль в деметилировании ДНК. Деметилирование ДНК также, как полагают, участвует в эксцизионной репарации нуклеотидов, обеспечиваемой с помощью Gadd45a (Barreto et al., 2007), хотя этот механизм всё ещё спорен (Jin et al., 2008).
Histone H3 lysine 9 methyltransferases
Пять гистоновых H3 lysine 9 (H3K9) methyltransferases идентифицировано у млекопитающих. Эти гистоновые methyltransferases (HMTs) имеют домен длиной ~130 аминокислот SET (Su(var), E(z), Trithorax) , ответственный за активность по метилированию лизина. K9 остаток модифицируется как mono- (K9me1), di- (K9me2) или trimethylation (K9me3). Suv39h1 и Suv39h2 являются H3K9 HMTs, которые преимущественно локализуются в DAPI-плотном гетерохроматиновом регионе (Aagaard et al., 1999). Suv39h1 или Suv39h2 нокаутные мыши обнаруживают нормальную жизнеспособность и плодовитость, но двойные мутанты рождаются не в соответствии с Менделевским распределением с задержкой роста и стерильностью самцов (Peters et al., 2001). Suv39h1/h2 двойные нулевые особи обнаруживают потерю H3K9me3 в перицентромерном гетерохроматине и неправильную регуляцию хромосом в эмбриональных фибробластах, это указывает на то, что их каталитические активности являются критическими для организации гетерохроматина (Peters et al., 2001). Известно также, что Suv39h1, вместе с белком retinoblastoma (Rb) и heterochromatin protein 1 (HP1), репрессируют гены, расположенные в эухроматине (Nielsen et al., 2002).
G9a (официально Ehmt2) является др. H3K9 HMT, которая распределяется в ядре за исключением перицентромерного гетерохроматина (Tachibana et al., 2001; Tachibana et al., 2002). Нокаут белка G9a в ES вызывает радикальную потерю H3K9me1 и H3K9me2 в эухроматине, указывая тем самым, что G9a является mono- и dimethyltransferase (Tachibana et al., 2002; Tachibana et al.,
2005). В исследованиях in vitro было показано, что G9a может также переносить три метильные группы на остаток лизина, хотя эта реакция m,jktt слабая, чем диметилирование (Patnaik et al., 2004). G9a ассоциирует с репрессорами транскрипции (Shi et al., 2003) и вносит вклад в транскрипционное молчание (Tachibana et al., 2002). В др. сообщении было показано, что G9a участвует также в транскрипционной активации независимо от HMT активности (Lee et al., 2006). G9a экспрессируется повсеместно в соматических тканях с высоким уровнем в семенниках, где G9a играет критическую роль в развитии зародышевых клеток (Tachibana et al., 2005; Tachibana et
al., 2007). G9a существенна также для раннего эмбриогенеза, поскольку G9a нокаутные мыши являются эмбриональными леталями (E9.5; Tachibana et al., 2002).
GLP, (G9a-like protein; официально Ehmt1), является H3K9 HMT, структурно сходен с G9a, формирует функциональный гетеродимер с G9a и распределяется в эухроматине. GLP нехватка достаточна, чтобы вызывать радикальную потерю H3K9me2 и реактивировать известные гены мишени для G9a (Tachibana et al., 2005). GLP нокаутные мыши погибают около E9.5 почти с идентичным фенотипом с тем, что у G9a (Tachibana et al., 2005). Однако паттерн экспрессии GLP
слегка отличен от такового для G9a, обнаруживая более высокую и более низкую экспрессию в ES клетках и семенниках, чем G9a, соотв. (Tachibana et al., 2005). Хотя только G9a, а не GLP присутствует в primordial germ cells (PGCs) около E7.5, эти клетки сохраняют H3K9me2 в ядрах (Seki et al., 2007). Т.о., эти белки могут функционировать независимо др. от др. в определенных условиях.
SETDB1/ESET является распределяющейся в эухроматине H3K9 HMT (Schultz et al., 2002). SETDB1 катализирует H3K9me2 и H3K9me3, а mAM, мышиный ATFa-associated factor AM
(официально Atf7ip), помогает SETDB1 превращать di- в trimethylation in vitro и в искусственной системе in vivo (Wang et al., 2003). SETDB1 участвует в замалчивании генов с транскрипционными репрессорам/корепрессорами, включая KAP-1, mAM, mSin3A/B (Schultz et al., 2002; Wang et al., 2003; Yang et al., 2003). Дефицит SETDB1 вызывает более тяжелый фенотип у мышей, чем таковой по Suv39h, G9a и GLP HMTs, и как результат пери-имплатационная летальность между E3.5 и 5.5, а нокаутные ES клетки не могут получены (Dodge et al., 2004).
Histone H3 lysine 27 methyltransferases Ezh2 является SET доменовым белком. катализирующим метилирование гистона H3 по лизину 27 (H3K27) (Cao et al., 2002). Ezh2, вместе с Eed, Suz12 и Rbbp4/7 формирует Polycomb repressive complex 2 (PRC2), который связан с инактивиацией X и кооперирует с PRC1, чтобы действовать как транскрипционный репрессор многих онтоегетических генов, включая homeobox гены (Schuettengruber et al., 2007). Дефицит или Eed или Suz12 вызывает резкое снижение Ezh2 и уровень метилирования H3K27, указывая, что Suz12 и Eed играют критическую роль в methyltransferase активности Ezh2 (Pasini et al., 2004; Montgomery et al., 2005). Интересно, что Eed нокаут вызывает потерю H3K27me1, K27me2 и K27me3, тогда как нокаут Suz12 ведет к редукции только K27me2 и K27me3, указывая, что Eed может вносить вклад в субстратную специфичность Ezh2 (Pasini et al., 2004; Montgomery et al., 2005). Ezh2 нокаутные мыши обладают пери-имплантационной летальностью (~E7.5), а мутантные ES клетки не могут быть получены из бластоцистов, указывая, что Ezh2 играет критическую роль в раннем развитии, при гаструляции (O'Carroll et al., 2001). Сходным образом как Suz12, так и Eed нокаутные мыши характеризуются эмбриональной летальностью около E7.5-8.5 (Faust et al., 1995; Pasini et al., 2004).
Т. о., исследования по всему геному выявили, что имеется множество PRC2 генов мишеней, которые перекрываются с H3K27me3 в геноме млекопитающих в стволовых и соматических клетках (Boyer et al., 2006; Bracken et al., 2006; Lee et al., 2006; Squazzo et al., 2006). G9a и GLP также обладают methyltransferase активностью в отношении H3K27, хотя она намного слабее, чем в отношении H3K9
in vitro (Tachibana et al., 2001; Tachibana et al., 2005). Дефицит G9a не меняет глобального уровня метилирования H3-K27 в ES клетках (Peters et al., 2003). Rougeulle et al. (2004) не выявили снижения метилирования H3K27 в
Xist гене на самцовой X-хромосоме при нокауте G9a ES клеток. Однако регионы мишени для G9a, в которых дефицит G9a вызывает гипометилирование ДНК, обнаруживают снижение уровней H3K9me2, также как и H3K27me2 в
G9a нокаутных ES клетках (Ikegami et al., 2007). Эти находки указывают на то, что G9a также катализирует H3K27me2
in vivo.
DNA methylation directs histone H3K9 methylation
Мутанты Arabidopsis нулевые по MET1, DNMT1-подобной CpG methyltransferase, обнаруживают кардинальную потерю метилирования H3K9 в гетерохроматиновых регионах, включая транспозоны (Tariq et al., 2003). В линии раковых клеток человека дефицит DNMT1 вызывает глобальное снижение H3K9me2 и H3K9me3, а трансфекция мышиной Dnmt1 устраняет этот фенотип (Espada et al., 2004). Интересно, что линия DNMT1 нокаутных клеток обнаруживает повышенную чувствительность к перевариванию с помощью micrococcal nuclease, указывая на роль DNMT1 в организации хроматина. Сходным образом нокаут DNMT1 с помощью siRNA вызывает снижение уровней H3K9me1, K9me2 и K9me3 в элементах межгенных спейсеров рибосомальной ДНК (rDNA) (Esteve et al., 2006). Однако Dnmt1/Dnmt3a/Dnmt3b трижды нокаутные ES клетки, которые обнаруживают полную потерю метилирования ДНК в некоторых повторяющихся последовательностях и импринтированных генах, не обнаруживая снижения глобальных уровней H3K9me2 и H3K9me3 ни изменения паттерна локализации H3K9me3 and HP1? (Tsumura et al., 2006).
Метилирование гистона H3K9/K27 управляет метилированием ДНК. Мутанты Neurospora crassa по генц, кодирующему Dim-5, H3K9 HMT, обнаруживают снижение уровней метилирования ДНК в специфических регионах, включая гены rDNA и транспозоны (Tamaru
and Selker, 2001). У Arabidopsis мутация H3K9 гена methyltransferase KRYPTONITE вызывает снижение уровней асимметричного метилирования CpNpG (где N может быть A, C, G или T) (Jackson et al., 2002). В ES клетках мышей двойные нулевые мутации Suv39h1 и Suv39h2 вызывают снижение уровней метилирования ДНК в основных сателлитных повторах перицентрического региона (Lehnertz et al., 2003). ES клетки, лишенные G9a обнаруживают снижение метилирования ДНК вместе с метилированием H3K9 в известном импринтируемом гене Snrpn (Xin et al., 2003). RNA interference EZH2 снижает уровни метилирования H3K27 и уровни метилирования ДНК в известных EZH2 генах мишенях (Vire et al., 2006).
SETDB1 может также влиять на метилирование ДНК поскольку SETDB1 взаимодействует с MBD1 и DNMT3A (Sarraf et al., 2004; Li et al., 2006a) (Discussed below). Однако нокаут SETDB1 в бластоцистах не вызывает изменений в уровнях метилирования ДНК во вкрапленных повторяющихся элементах (Dodge et al., 2004).
Анализ метилирования ДНК по всему геному клеток ES показал, что G9a дефицит вызывает снижение уровней метилирования ДНК на 1.6% (32 loci) из общего количества ~2,000 проанализированных локусов (Ikegami et al., 2007). В этих регионах нокаут G9a в ES клетках ведет к снижению уровней H3K9me2 и/или H3K27me2, а G9a-трансген восстанавливает статус метилирования ДНК в G9a нокаутных клетках. Важно, однако, что уровни метилирования ДНК остаются и нтактными для большинства генетических локусов при нокауте G9a
в ES клетках. Следовательно, G9a участвует в метилировании ДНК локус-специфическим образом (Ikegami et al., 2007). Всё это указывает на то, что метилирование H3K9 и K27 может управлять метилированием ДНК из генных регионов, показывая как происходит локальное метилирование или деметилирование ДНК, когда Dnmts, глобальные регуляторы, останавливают по всему геному профиль метилирования ДНК.
Interaction between H3K9/27 HMTs and Dnmts
Исследования ко-экспрессии выявили, что SUV39H1 человека соединяется с мышиными Dnmt1 и Dnmt3a (Fuks et al., 2003a). В линии клеток рака груди последовательный ChIP анализ с антителами против DNMT3A и SETDB1 показал, что SETDB1 соединяется с DNMT3A на хроматине (Li et al., 2006a). Прямое взаимодействие между H3K27 methyltransferase EZH2 и DNMTs (DNMT1, 3A и 3B) (Vire et al., 2006), подтверждает мнение, что метилирование H3K27 связано с метилированием ДНК. Более того, DNMTs ассоциируют с EZH2-target генами в присутствии EZH2 (Vire et al., 2006).
G9a также взаимодействует с DNMT1 и ко-локализуется специфически в синхронизированных в S-фазе клетках человека (Esteve et al., 2006). В сообщении и G9a и DNMT1 локализовались на сайтах репликации, на что указывает строгое включение BrdU. Следовательно, G9a и DNMT1 могут кооперативно поддерживать метилирование H3K9 и паттерн метилирования ДНК в фокусах репликации. Молекулярное взаимодействие между белками, непосредственно участвующими в метилировании ДНК и H3K9/K27 указывает на то, что обе модификации могут быть базой для рекрутирования других.
Proteins recognizing DNA methylation
Геном человека кодирует 12 белков, содержащих methyl-CpG-binding domain (MBD), который является белковым мотивом, ответственным за соединение с метилированными CpG динуклеотидами (http://www.ebi.ac.uk/interpro). Среди них, MeCP2, MBD1, MBD2 и MBD4 специфически распознают methylCpG (Klose and Bird, 2006). В противовес животным, дефицитным по Dnmts и HMTs, мутанты по каждому из этих 4 MBD белков доживают до взрослого периода (Fatemi and Wade, 2006). Однако мутация X-сцепленного гена MeCP2 вызывает нейрологическое заболевание Rett syndrome почти исключительно у женщин, который приводит к аресту развития в возрасте между 6 и 18 мес. у человека (Amir et al., 1999).
Kaiso, (официально Zbtb33), Zbtb4 и Zbtb38 используют родоначальный three-zinc-finger мотив, чтобы связываться с methylCpG, но дифференциально экспрессируются в тканях мышей, подтверждая неперекрываемость функций или мишеней (Prokhortchouk et al., 2001; Filion et al., 2006).
UHRF1 (ubiqutin-like, containing PHD and RING finger domains
1), Dnmt1 партнер по связыванию, преимущественно соединяется с полу-метилированной ДНК посредством своего SRA (SET и RING ассоциированного) домена (Bostick et al., 2007). Нокаут Uhrf1 гена ведет к неправильной локализации Dnmt1 в S фазе и снижению уровней метилирования ДНК, подтверждая роль UHRF1 в поддержании паттернов метилирования ДНК (Bostick et al., 2007; Sharif et al., 2007).
Proteins recognizing histone methylation
Chromodomain распознает метилирование лизинов 9 и 27, которые располагаются внутри высоко родственных последовательностей мотива Ala-Arg-Lys-Ser (ARKS). HP1 белки (HP1α,β и γ) метилируют H3K9 посредством своих chromodomains (Lachner et al., 2001). HP1 также непосредственно соединяется с SUV39H1 (Aagaard et al., 1999) и G9a (Sampath et al., 2007). Др. chromodomain белок, Polycomb (Pc), член PRC1, соединяется с метилированным H3K27 (Fischle et al., 2003). Биохимические опыты и кристаллография продемонстрировали, что Drosophila HP1 и Pc специфически взаимодействуют с H3K9me3 и H3K27me3, соот. (Fischle et al., 2003). Однако у мышей ни один из 5 гомологов Polycomb (Cbx2, Cbx4, Cbx6, Cbx7 и Cbx8) не обнаруживает предпочтения к K27me3 (Bernstein et al., 2006). Вместо этого, Cbx2 и Cbx7 обнаруживают строгое сродство к K9me3 и K27me3, а Cbx4 напротив предпочитает K9me3 подобно HP1 (Bernstein et al., 2006). HP1 и Cbx4 могут выполнять роль по дискриминации метилирования H3K9 от H3K27 у млекопитающих.
Tudor домены, которые структурно родственны chromodomain, соединяются с methyllysines. JMJD2A распознает H3K4/H3K20me3 посредством домена и также деметилирует H3K9/H3K36me3 посредством jumonji C (jmjC) домена (Huang et al., 2006; Klose et al., 2006). Белки, содержащие MBT (malignant brain tumor) домен, который также сходен с chromodomain, имеет широкий спектр мишеней лизинов, включая H3K4, H3K9, H3K27, H3K36, H4K20 и K26 из гистона H1 изотипа b (Trojer and Reinberg, 2008). Др. тип, PHD (plant homeodomain) finger домен не является структурно родственным chromodomain. Различные PHD finger белки участвуют в активации транскрипции, репрессии и рекомбинации, они распознают H3K4me3 (Kouzarides et al.,'2007). Недавно сообщалось, что SMCX (официально JARID1C), jmjC домен-содержащая H3K4me3 demethylase связывает H3K9me3 посредством PHD домена (Iwase et al., 2007).
Proteins connecting DNA methylation and histone methyla-
tion
MBD белки играют роль в связывании метилирования ДНК с метилированием гистонов. MBD1 непосредственно взаимодействует с Suv39h1 in vitro и ко-локализуется с Suv39h1 in vivo MBD-зависимым способом (Fujita et al., 2003). MBD1 взаимодействует также с HP1 (Fujita et al., 2003). Сходным образом, MeCP2, как было установлено, ассоциирует с H3K9 methyltransferase активностью, хотя точный партнер(ы) не идентифицирован (Fuks et al., 2003b). Следовательно, MBD белки играют роль в рекрутировании метилирования H3K9 на сайт метилирования ДНК и/или HMT рекрутирует MBD белки, чтобы облегчить их связывание с метилированными CpG. Недавно было продемонстрировано in vivo взаимодействие MBD1 с SETDB1 (Sarraf et al., 2004). Во время репликации chromatin assembly factor-1 (CAF-1,
официально CHAF1), histone chaperone комплекс, взаимодействующий с PCNA, соединяются, чтобы сформировать специфичный для S-фазы MBD1/SETDB1/CAF-1 комплекс (Sarraf et al., 2004). MBD1 может участвовать в поддержании паттерна метилирования H3K9 одновременно с метилированием ДНК в фокусах репликации. Наконец, поскольку SETDB1 сам обладает MBD
мотивом, то внутренний MBD может управлять метилированием H3K9 в CpG метилированных геномных регионах.
HP1 белки сцеплены с системой метилирования ДНК. HP1 белки взаимодействуют с DNMT1, DNMT3A и DNMT3B
in vitro или при анализе ко-экспрессии (Fuks et al., 2003a; Smallwood et al., 2007).
In vivo, HP1? взаимодействует с Dnmt3b, но не взаимодействует или очень слоабо взаимодействует с Dnmt1 и Dnmt3a (Lehnertz et al., 2003). Следовательно, HP1 может рекрутировать Dnmts, где метилируется H3K9. HP1 белки, по-видимому, локализуются дифференциально в ядре во время клеточного цикла. Напр., HP1? локализуется наиболее предпочтительно среди трех HP1s на центромерах во время интерфазы, тогда как только HP1? локализуетя на центромерах во время метафазы в клетках человека (Hayakawa et al., 2003). Т.о., HP1 может влиять на локализацию Dnmt во время клеточного цикла. HP1 белки также непосредственно взаимодействуют с MeCP2 в экспериментах по избыточной экспрессии (Agarwal et al., 2007). Эти результаты указывают на то, что метилирование H3K9 рекрутирует метилирование ДНК посредством HP1 белков и что локализация HP1 испытывает влияние со стороны паттерна метилирования ДНК посредством белков MBD.
DNA methylation and other histone modifications
У млекопитающих существует, по крайней мере, 10 известных или предполагаемых H3K4
methyltransferases, которые в целом распределены по MLL (mixed lineage leukemia) семейству, Set1 семейству и др. (Ruthenburg et al., 2007). MLL, a H3K4 HMT, является членом группы trithorax, которая в целом функционирует антагонистически против белков группы polycomb. На ген мишень trithorax/polycomb, Hoxc8, дефицит MLL вызывает снижение экспрессии и уровней метилирования H3K4 вместе с повышением уровня метилирования ДНК в эмбриональных фибробластах мышей (Milne et al., 2002). Сходным образом. мыши, лишенные SET домена в MLL обнаруживают пониженные H3K4me1 и повышенные уровни метилирования ДНК в гене Hoxd4 (Terranova et al., 2006). Интересно, что этот мутант не обнаруживает глобальных изменений метилирования H3K4me1 и метилирования ДНК, указывая, что метилирование H3K4 затрагивает метилирование ДНК DNA локус-специфическим способом. Это мнение подтверждается недавним исследованием генома, показавши, что имеется существенное количество генов, маркированных с помощью H3K4me3 с ДНК гипер- или гипометилировнием промоторов (Fouse et al., 2008). Примечательно, что MLL обладает гомологией с Dnmt1 в methyltransferase домене, который избирательно соединяется в неметилированными CpG последовательностями in vitro (Birke et al., 2002). Однако форсированная экспрессия экзогенного MLL в MLL нокаутных клетказ не снижала уровней метилирования ДНК (Milne et al., 2002). Следовательно, метилирование H3K4 может защищать определенные локусы от метилирования ДНК.
Histone deacetylase 1 (HDAC1) ассоциирует с MeCP2 в корепрессорном комплексе, представляя модель, в которой локусы, с гиперметилированными CpGs, подвергаются деацетилированию гистонов с помощью этого комплекса (Nan et al., 1998). Кроме того, HDAC1 и HDAC2 непосредственно взаимодействуют с DNMT1 (Fuks et al., 2003b; Robertson et al., 2000; Rountree et al., 2000). Обработка
Neurospora crassa Trichostatin A (TSA), ингибитором histone deacetylase, вызывает снижение метилирования ДНК в некоторых генах, тогда как она не влияет на глобальное метилирование ДНК в повторяющихся последовательностях (Selker, 1998). В линии клеток колоректальной карциномы человека обработка с помощью TSA не влияет на статус метилирования ДНК CpG островков из двух молчащих генов, которые были тяжело метелированы (Cameron et al., 1999). TSA обработка снижает уровни мРНК и белка DNMT3B в эндометриальных клетках человека и их уровни DNMT1 в Jurkat T клетках, соотв. (Xiong et al., 2005; Januchowski et al., 2007). Следовательно, по-видимому, в каждом случае существует и зависимость между ациетилированием гистонов и метилированием ДНК.
Molecular link among DNA methylation, chromatin organization and non-coding RNAs
Chromatin remodeling factors, linker histone and DNA methylation
Помимо гистоновых модификаций некоторые белки, ассоциирующие с хроматином, связаны с метилированием ДНК (Fig. 2). Один из них Lsh (lymphoid specific helicase, официально
Hells), член семейства SNF2 ATPase/helicase, который участвует в ремоделировании хроматина. Ткани от Lsh нокаутных мышей обнаруживают гипометилирование ДНК по сателлитным и вкрапленным повторяющимся последовательностям, а также по уникальным последовательностям, включая ткане-специфичные гены, beta-Globin и Pgk-2 (Dennis et al., 2001). Lsh
взаимодействует с Dnmt3a и Dnmt3b, но не с Dnmt1 (Zhu et al., 2006). Деметилирование ДНК по повторяющимся последовательностям, выглядит более тяжелым в Lsh нокаутных клетках, чем таковое в двойных нокаутных Dnmt3a/Dnmt3b клетках, указывая тем самым, что активность Dnmt1 также испытывает влияние со стороны Lsh (Dennis et al., 2001). Dnmts могут нуждаться в функции ремоделирования хроматина Lsh для своих каталитических активностей.
ATRX (X-linked alpha thalassemia/mental retardation) др. член семейства SNF2 белков ATPase/helicase. Пациенты с мутациями в гене ATRX характеризуются гипометлирование ДНК и гипометилированием сателлитных повторов (Gibbons et al., 2000). Белок ATRX взаимодействует с MeCP2 в ко-трансфицированных клетках и оба белка ко-локализуются в домене DAPI-плотного гетерохроматина в головном мозге взрослых мышей (Nan et al., 2007). В MeCP2-нулевом головном мозге ATRX делокализуется из хроматина (Nan et al., 2007). Это указывает на то, что метилирование ДНК и ATRX ремоделирование хроматина влияют др. на др.
Несколько сообщений продемонстрировали, что линкерный гистон H1 преимущественно соединяется с метилированной ДНК (Levine et al., 1993; McArthur and Thomas, 1996), тогда как в др. сообщалось, что метилирование ДНК не влияет на связывание H1 (Campoy et al., 1995; Nightingale and Wolffe, 1995). Недавно FRAP анализ показал, что двойной нокаут Dnmt3a/Dnmt3b вызывает задержку кинетики восстановления линкерных гистонов, H1 и H5, в ES клетках (Gilbert et
al., 2007). ДНК метилирование, по-видимому, облегчает сборку линкерных гистонов in vivo.
Напротив, гистон H1 влияет на статус метилирования ДНК также. Нокаут по трем изотипам H1 (H1c, H1d, and H1e) вызывает глобальное изменение структуры хроматина, индуцирует деметилирование ДНК по специфическим генам,но не в сателлитных или вкрапленных повторяющихся последовательностях (Fan et al., 2005). Этот мутант обнаруживает также глобальное снижение ацетилирования H3K12 и H3K27me3.
Итак, белки, ассоциированные с хроматином влияют на установление паттерна метилирования ДНК прямо или косвенно посредством модификаций гистонов и наоборот, метилирование ДНК влияет на распределение и/или функцию этих белков.
Higher-order chromatin and DNA methylation
Метилирование ДНК играет роль в организации структуры хроматина высшего порядка. Dnmt3b-дефицитные эмбриональные фибробласты мышей обнаруживают пониженные уровни метилирования ДНК на эндогенных повторяющихся последовательностях , высокую частоту полиплоидии и анеуплоидии в культуре (Dodge et al., 2005). Lymphoblastoid линия клеток, полученная от пациентов с синдромом иммунодефицита, нестабильностью центромерной области и лицевыми аномалиями (ICF синдром), который вызван мутациями гена
DNMT3B , также обнаруживает хромосомные аномалии, включая деконденсацию метафазных хромосом, образование мостиков в анафазном и/или интерфазном хроматине и образованием аномальных петель хроматина (Gisselsson et al., 2005). Dnmt3a/Dnmt3b двойные нулевые ES обнаруживают повышенное образование кластеров перицентромерного гетерохроматина в немногие крупные хромоцентры (Gilbert et al., 2007). Следовательно, метилирование ДНК вносит вклад в динамику хромосом. Однако тройной нокаут Dnmt1, Dnmt3a и Dnmt3b не влияет на количество хромосом в ES клетках мышей (Tsumura et al., 2006).
Non-coding RNA in the directing of DNA methylation and
histone modification
генетический анализ растений показал, что некодирующая РНК (ncRNA) участвует в метилировании ДНК и эта система известна как РНК-зависимое метилирование ДНК (Zaratiegui et al., 2007). В клетках человека трансфекция 21-nucleotide (nt) РНК гомологов в промоторную последовательность гена
EF1A увеличивала уровень метилирования ДНК в промоторной области (Morris et al., 2004). В гене
RASSF1A short hairpin RNAs (shRNA) , содержащая 21-nt
последовательность, комплементарную промоторному CpG островку, индуцирует очень низкий уровень или не выявляемое метилирование ДНК в области мишени в зависимости от метода анализа (Castanotto et al., 2005). Недавно нокаут Piwi белков, MIWI2 и MILI, которые соединяются с
26-31-nt piRNAs (Piwi-interacting small RNAs), вызывал снижение уровней метилирования ДНК транспозонов в зародышевых клетках самцов мышей (Aravin et al., 2007; Kuramochi-Miyagawa
et al., 2008). Эти находки указывают на вовлечение piRNA в систему метилирования ДНК. Напротив, имеются сообщения, что ncRNA не вызывают метилирования ДНК. Последовательность в19-nt, перекрывающая точку старта транскрипции в гене
PGR человека снижает уровень мРНК, но не индуцирует метилирования ДНК вокруг сайта мишени в линии раковых клеток человека (Janowski et al., 2005). Сходным образом, 21nt double-stranded RNAs (dsRNAs), которые гомологичны CpG островку промотра CDH1, индуцирует деметилирование H3K9me2 и транскрипционное молчание, но не метилирование ДНК (Ting et al., 2005). Более того, РНК-обусловленная репрессия транскрипции осуществляется в клетках, генетически лишенных DNMT1 и DNMT3B (Ting et al., 2005). Итак, РНК-зависимая индукция метилирования ДНК всё ещё остается дискуссионной у млекопитающих. Как отмечалось выше, shRNA-трансфицированные клетки, обнаруживают повышенные уровни H3K9me2 в CHD1 промоторе (Ting et al., 2005). Сходным образом, 21 nt РНК гомологи, интегрированные с промотором гена
CCR5, индуцируют H3K9me2 в промоторе (Kim et al., 2006). Уровни H3K27me3 и EZH2 увеличиваются в промоторе гена
RASSF1A в клетках HeLa, стабильно экспрессирующих shRNA targeting the promoter (Kim et al., 2006). Взаимодействие между длинной ncRNA и G9a или EZH2 установлено недавно (Nagano et al., 2008; Pandey et al., 2008). Эти сообщения указывают на то, что ncRNA играет роль в системе метилирования H3K9 и K27 у млекопитающих. В противовес выше приведенным находкам было предположено, что ncRNAs запускак деметилирование ДНК и H3K9 в системах млекопитающих. Линия раковых клеток человека, трансфицированная 21-nt dsRNA, нацеленной на промотор E-cadherin, индуцирует транскрипцию и снижает уровни H3K9me2 и H3K9me3 в области мишени с CpG островком (Li et al., 2006b). Imamura et al., (2004) сообщили, что трансфекция 0.2-1.0 kbp антисмысловой ncRNAs, соответствующей CpG островку гена
Sphk1 индуцирует деметилирование CpG сайтов и метилирование CC(A/T)GG сайтов в T-DMR в CpG островке. Важно, что эти антисмысловые РНК фактически транскрибируются с CpG островка в тканях крыс (Imamura et al., 2004).
Conclusion
The cell- and tissue-specific expression of genes, including
transcription factors, have traditionally been explained through the
regulation by the transcription factors active in the cell/tissue type.
However, the regulation by transcription factors is generally tem-
porally constrained and rapidly changeable in response to environ-
mental and extra-cellular stimuli. It is becoming clear that various
genes are controlled by epigenetic systems of DNA methylation
and histone modifications. DNA methylation profiles are, in a
certain sense, the epigenetic memory that is indispensable for cells
and tissues to maintain their unique features.
Analysis of DNA methylation profiles provides us a survey of
the epigenetic status of the entire genome, because various
histone modifications and chromatin remodeling generally reflect
the genome-wide DNA methylation status (Fig. 2). We are now in
the era of the epigenome, the “Total information of epigenetic
marks on the genome”. The epigenome connects the genome
(DNA sequence) and transcriptome by introducing another layer
of genetic control: a stable memory of the genetic activity for
numerous genes, which is heritable to the next generation. The
epigenome will provide new insight into mammalian development
and the differentiation of cells. In addition, epigenetic analysis
provides a new paradigm for understanding the normal and
abnormal status of cells. Direct/indirect interactions among Dnmts,
histone modification enzymes, histone subtypes, chromatin re-
modeling factors and ncRNAs underlie the establishment of the
cell-type specific epigenome.
Сайт создан в системе
uCoz  Fig. 1. Cell-type-specific DNA methylation profiles consisting of differentially methylated T-DMRs. Each cell type (ES cells and TS cells) has a unique pattern of DNA methylation at CpGs within T-DMRs (lower panel). These differential methylation status in T-DMRs constitute cell-type-specific DNA methylation profiles genome-wide (upper panel). The DNA methylation profile of a cell type can be used as a novel tool to define and characterize the cell type.
Fig. 1. Cell-type-specific DNA methylation profiles consisting of differentially methylated T-DMRs. Each cell type (ES cells and TS cells) has a unique pattern of DNA methylation at CpGs within T-DMRs (lower panel). These differential methylation status in T-DMRs constitute cell-type-specific DNA methylation profiles genome-wide (upper panel). The DNA methylation profile of a cell type can be used as a novel tool to define and characterize the cell type.  Fig. 2. Molecular link between DNA methylation, chromatin structure and histone modifications in inactive (A) and active (B) genomic regions. BER, base excision repair proteins (e.g. AP endonuclease, DNA polymerase I); Chromo, chromodomain proteins (e.g. HP1, Pc); DNMTs, DNA methyltransferases (e.g. Dnmt1, Dnmt3s); HATs, histone acetylases (e.g. CBP/P300); HDACs, histone deacetylases (e.g. HDAC1, HDAC2); MBT, MBT domain proteins; MeC-BPs, methylCpG binding proteins (e.g. MBDs, MeCP2, UHRF1); PHD, PHD finger proteins (e.g. BPTF, SMCX); TDG, thymine DNA glycosylase; Tudor, Tudor domain
proteins.
Fig. 2. Molecular link between DNA methylation, chromatin structure and histone modifications in inactive (A) and active (B) genomic regions. BER, base excision repair proteins (e.g. AP endonuclease, DNA polymerase I); Chromo, chromodomain proteins (e.g. HP1, Pc); DNMTs, DNA methyltransferases (e.g. Dnmt1, Dnmt3s); HATs, histone acetylases (e.g. CBP/P300); HDACs, histone deacetylases (e.g. HDAC1, HDAC2); MBT, MBT domain proteins; MeC-BPs, methylCpG binding proteins (e.g. MBDs, MeCP2, UHRF1); PHD, PHD finger proteins (e.g. BPTF, SMCX); TDG, thymine DNA glycosylase; Tudor, Tudor domain
proteins.